Nat. Methods | 基于最优传输的空间转录组细胞命运景观学习
Nat. Methods | 基于最优传输的空间转录组细胞命运景观学习

DrugAI
发布于 2026-01-06 13:42:06
发布于 2026-01-06 13:42:06
DRUGONE
在发育等动态生物过程中,空间转录组技术正在革新对组织空间组织机制的研究。如何从多个时间点的空间转录组数据中推断细胞命运轨迹已成为关键问题,需要新的计算方法。研究人员提出 STORIES,一种基于最优传输(Optimal Transport, OT)的新方法,用以学习具有空间信息的分化势能。STORIES 扩展了 OT 框架,使用 Fused Gromov–Wasserstein(FGW) 距离作为损失函数,从时间序列空间转录组数据中学习细胞分化的因果模型。研究人员在三套大规模 Stereo-seq 空间时间图谱(小鼠发育、斑马鱼发育、墨西哥钝口螈再生)中验证了该方法,显示 STORIES 在空间一致性与生物解释性上均优于现有方法,并能恢复神经再生与胶质生成中的已知基因标志物与潜在驱动因子。

空间转录组技术能够揭示组织内细胞的空间分布及基因表达模式。随着 Stereo-seq 等高分辨技术的出现,研究人员可以在单细胞水平追踪发育或疾病进程中细胞的时空动态。
传统轨迹推断方法(如 Monocle、RNA velocity)主要依赖时间或剪接速率,难以捕捉空间信息。基于最优传输的模型能够刻画细胞间的转变概率或势能变化,为描述分化过程提供了数学基础,但这些方法通常无法有效整合空间坐标,因为组织切片在不同时间点会发生旋转、平移甚至形态变化。
研究人员因此发展出 STORIES,使其能在 FGW 框架下利用空间距离矩阵,而无需对齐切片,从而学习空间一致的分化势能。这一方法自然继承了 Waddington 表观遗传景观的理念,将细胞分化视为势能最小化的动态过程。
方法概述
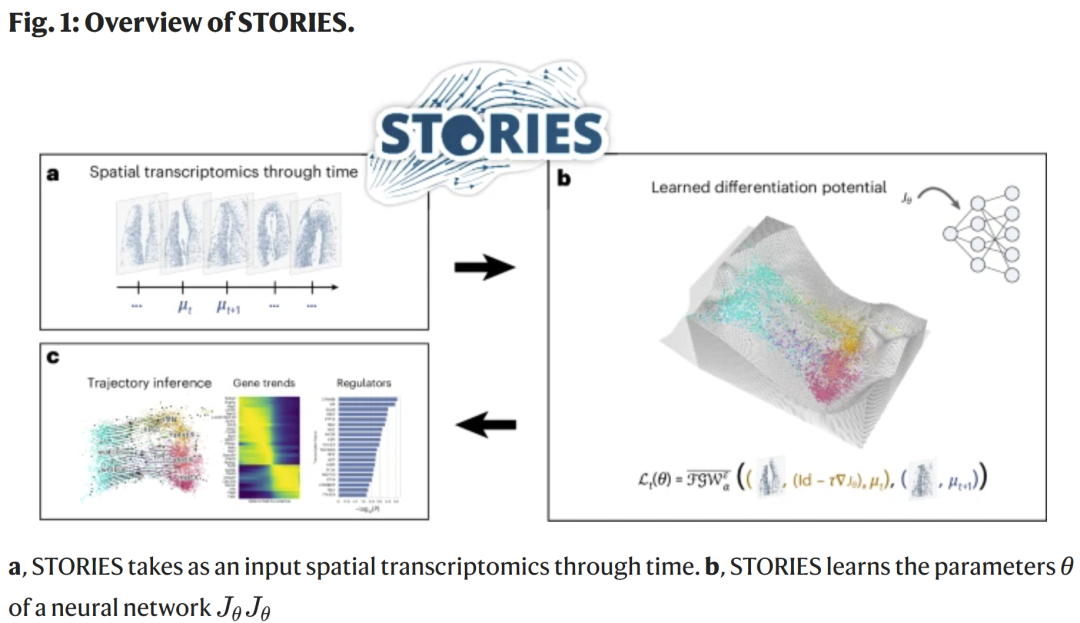
STORIES 接收多个时间点的空间转录组切片数据输入。神经网络 学习将每个细胞的基因表达映射到一个势能值,用以描述分化潜能。势能的梯度提供细胞状态演化方向,势能本身则可替代伪时间,用于基因趋势与调控因子的研究。
不同于线性 OT,STORIES 使用 FGW 距离来比较时间点间的细胞分布,使模型对旋转、平移等形变保持不变。
此外,该框架通过时间序列数据学习连续的细胞动力学模型,并能预测未来时间点的细胞表达状态。模型在 GPU 上的训练可在20分钟内完成数十万细胞的训练任务。STORIES 提供开源 Python 实现,兼容 Scverse、Scanpy、CellRank 等工具,便于数据预处理与可视化分析。
结果
STORIES 方法性能验证
研究人员在三组大规模 Stereo-seq 数据集上对 STORIES 进行了基准测试:
- 小鼠发育数据集
- 斑马鱼发育数据集
- 钝口螈脑再生数据集
结果显示:
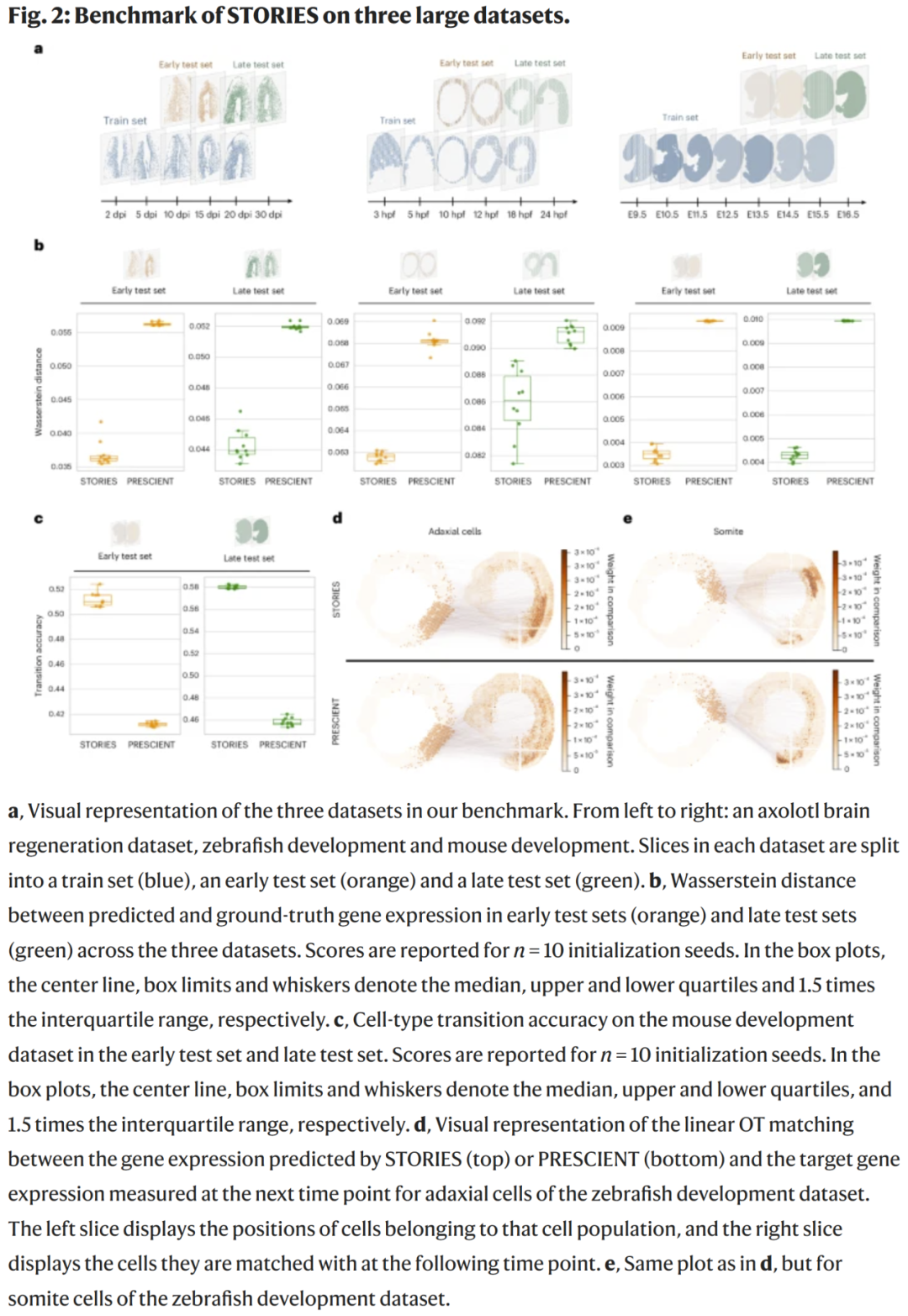
- 相较于仅使用线性 OT 的版本(α=0),STORIES 通过 FGW 融合空间信息后,能更准确预测基因表达的演化趋势。
- 与现有最优传输方法 PRESCIENT 比较,STORIES 在 Wasserstein 距离与细胞类型转移准确率上均显著优于对照方法。
- 视觉化结果表明,STORIES 能在斑马鱼发育中正确匹配体节细胞与相应时间点的同类细胞,而传统方法在空间映射上出现明显偏差。
这些结果表明,引入空间信息能有效提高轨迹推断的生物学一致性。
STORIES 揭示钝口螈神经再生的驱动因子
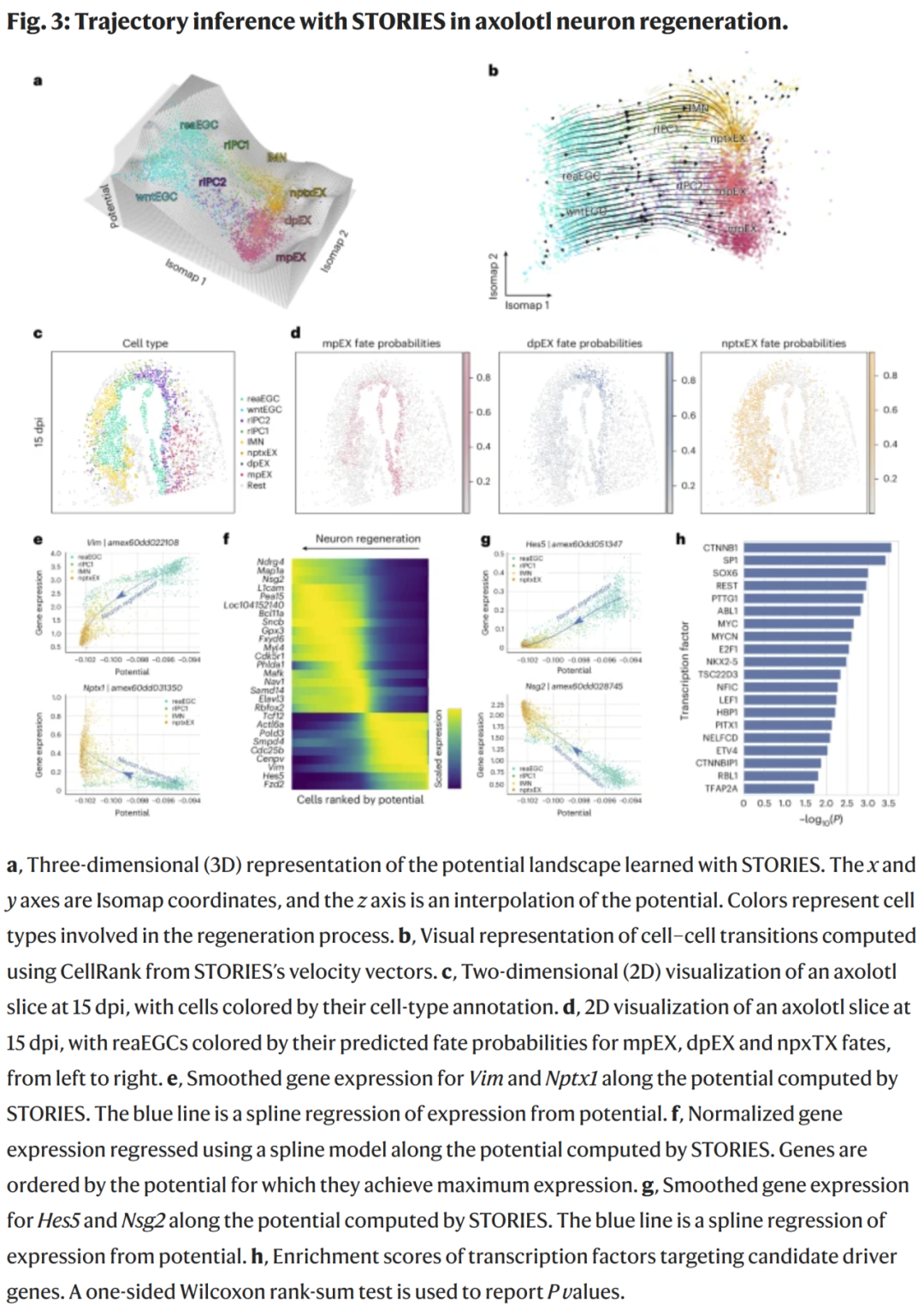
研究人员在钝口螈脑再生的空间转录组数据中训练 STORIES,聚焦于神经再生相关的细胞类型,包括 EGC、rIPC、IMN 及不同类型的兴奋性神经元。
STORIES 学得的势能景观显示,EGCs 与 rIPCs 处于高势能(前体状态),而 NptxEX、dpEX、mpEX 等成熟神经元处于低势能状态。
通过 CellRank 对势能梯度计算细胞转移,发现 STORIES 自动恢复出三个主要的分化通路:
- wntEGC → mpEX
- reaEGC → rIPC2 → dpEX
- reaEGC → rIPC1 → IMN → nptxEX
这一结果无需手动设定起始点或空间区域。
进一步分析发现,空间位置对命运决策存在影响:损伤右侧的 reaEGCs 更倾向分化为 mpEX,而左侧的 reaEGCs 更倾向生成 nptxEX(图3c–d)。
基因趋势分析揭示:
- Vim 随分化降低,Nptx1 随分化上升。
- 新发现的驱动候选基因包括 Hes5、Cdc25b、Map1a、L1cam 以及较少研究的 Nsg2。
转录因子富集分析表明,CTNNB1、SP1、MYC、SOX6、REST 等在神经再生中具有潜在调控作用。
综上,STORIES 不仅重建了空间-分化景观,还揭示了关键调控网络。
STORIES 识别小鼠中脑胶质生成的调控机制
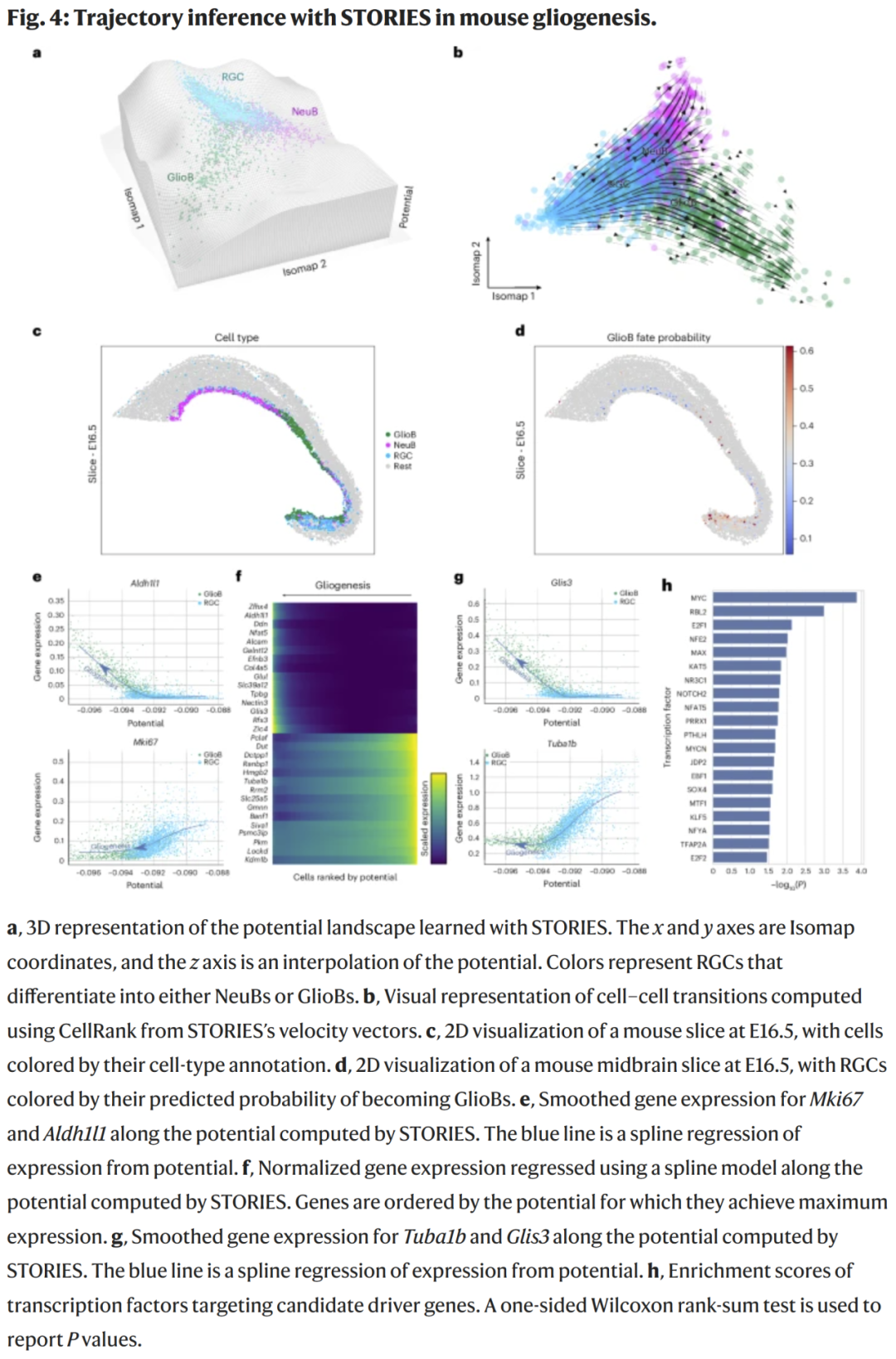
研究人员进一步在小鼠中脑发育数据中测试 STORIES。
结果显示:
- 势能景观正确地将放射状胶质细胞(RGCs)置于高势能区,而将神经母细胞(NeuBs)和胶质母细胞(GlioBs)置于低势能区。
- 基于势能梯度计算的细胞转移关系揭示出 RGC 向神经元和胶质细胞的分化分支。
- 在空间层面,RGC 的分化命运与位置相关:前端 RGC 倾向生成神经元,后端 RGC 倾向生成胶质细胞。
- 基因趋势分析验证了已知标志物变化:Mki67(增殖标志物)下降,Aldh1l1(星形胶质细胞标志物)上升。
- 候选驱动基因包括早期的 Gmnn、Rrm2、Hmgb2、Tuba1b,以及晚期的 Glul、Glis3。
- 富集分析指出关键转录因子如 SOX4、NOTCH2、MYC、MYCN、MAX 可能调控胶质生成过程。
讨论
空间转录组技术的迅猛发展为动态生物过程(如发育与疾病进展)的系统解析提供了新的契机。STORIES 的提出为分析时间序列空间单细胞数据提供了统一、可解释的数学模型。
通过在多个数据集的基准评估与案例分析,研究人员证明:
- 将空间信息纳入轨迹推断显著提升预测精度;
- 势能模型提供了分化过程的自然解释框架,可同时揭示空间模式与调控网络。
STORIES 的创新之处在于其能学习空间敏感的势能函数,使得在形态变化显著的发育过程也能保持空间一致性。未来方向包括:
- 将模型拓展至能预测细胞迁移路径的空间能量函数;
- 引入细胞间相互作用能量以探索形态发生与免疫反应的集体动力学;
- 结合 Gromov–Wasserstein 几何与反应-扩散模型,以更全面地刻画生物系统的自组织与再生机制。
整理 | DrugOne团队
参考资料
Huizing, GJ., Samaran, J., Capocefalo, D. et al. STORIES: learning cell fate landscapes from spatial transcriptomics using optimal transport. Nat Methods (2025).
https://doi.org/10.1038/s41592-025-02855-4
内容为【DrugOne】公众号原创|转载请注明来源
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-11-04,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读

