(IF=48.8)顶刊杂志的带有超详细python单细胞代码和数据学习的文献分享
(IF=48.8)顶刊杂志的带有超详细python单细胞代码和数据学习的文献分享
生信技能树
发布于 2025-11-20 11:34:12
发布于 2025-11-20 11:34:12
今天来学习一篇发表在 Cancer Cell 杂志(IF=48.8)上的文献,于2022年12月12日发表,标题为《High-resolution single-cell atlas reveals diversity and plasticity of tissue-resident neutrophils in non-small cell lung cancer》,通讯作者Andreas Pircher,Zlatko Trajanoski。
这篇文献是一篇极其典范的单细胞python流派的数据分析文章,集合了单细胞数据预处理,数据合并,数据整合,数据注释等工作。跨数据集和大数据量!应该有很多细节值得学习,更特别的是注释工作,做这样的一个大数据量的图谱类细胞类型注释,可以看看里面到底是如何处理各种分析细节的!
数据和代码
文章的数据队列,基本上都可以下载。提供的数据在这里:
Processed input data:https://doi.org/10.5281/zenodo.6411867
可复现的代码:https://github.com/icbi-lab/luca
提供了清晰的分析步骤:

下游分析:

代码一撇:
看一个脚本:
# ---
# jupyter:
# jupytext:
# formats: ipynb,py:light
# text_representation:
# extension: .py
# format_name: light
# format_version: '1.5'
# jupytext_version: 1.6.0
# kernelspec:
# display_name: Python [conda env:.conda-scanpy_2020-12]
# language: python
# name: conda-env-.conda-scanpy_2020-12-py
# ---
import scanpy as sc
import pandas as pd
from glob import glob
from pathlib import Path
import re
import scipy.sparse
from multiprocessing import Pool
import anndata
filenames = glob("../../data/11_own_datasets/batch1_3patients/processed/*.csv")
meta = pd.read_excel("../../tables/patient_table_batch1_3_patients.xlsx", engine="openpyxl", skiprows=1, skipfooter=1)
meta.rename(columns={"Tumornummer": "tumor_id", "Patient": "patient", "Alter": "age", "Geschlecht": "sex", "Tumor": "tumor_type"}, inplace=True)
meta["sex"] = [{"W": 'f', "M":'m'}[x] for x in meta["sex"]]
def load_counts(filename):
p = Path(filename)
((patient, tissue), ) = re.findall("(P\d+)_(.*)\.csv", p.name)
expr = pd.read_csv(p, skiprows=5, index_col=0)
gene_expr = expr.loc[(~expr.index.str.contains("\(Ab\)") & ~expr.index.str.startswith("Lex_")), :]
ab_expr = expr.loc[expr.index.str.contains("\(Ab\)"), :]
obs = pd.DataFrame().assign(cell_id=expr.columns)
obs["patient"] = patient
obs["tissue"] = tissue
obs.set_index("cell_id", inplace=True)
adata = sc.AnnData(X=scipy.sparse.csc_matrix(gene_expr).T, obs=obs)
adata.var_names = gene_expr.index
adata.obsm["surface_protein"] = ab_expr.T
return adata
with Pool(16) as p:
adatas = p.map(load_counts, filenames)
adata = anndata.concat(adatas, index_unique="_", join="outer")
meta["patient"] = [x.strip() for x in meta["patient"]]
adata.obs = adata.obs.reset_index().merge(meta, on=["patient"], how="left").set_index("cell_id")
adata
adata.obs.drop_duplicates()
adata.obs["condition"] = "NSCLC"
adata.obs["origin"] = ["tumor_primary"if c == "Tumor"else"normal_adjacent"for c in adata.obs["tissue"]]
adata.obs["sample"] = [f"{patient}_{origin}"for patient, origin in zip(adata.obs["patient"], adata.obs["origin"])]
adata.obs["sex"] = [{"m": "male", "f": "female"}[s] for s in adata.obs["sex"]]
adata.obs["tissue"] = "lung"
adata.obs.drop_duplicates()
# !mkdir -p "../../data/11_own_datasets/batch1_3patients/h5ad_raw"
adata.write_h5ad("../../data/11_own_datasets/batch1_3patients/h5ad_raw/batch1_3patients.h5ad", compression="lzf")
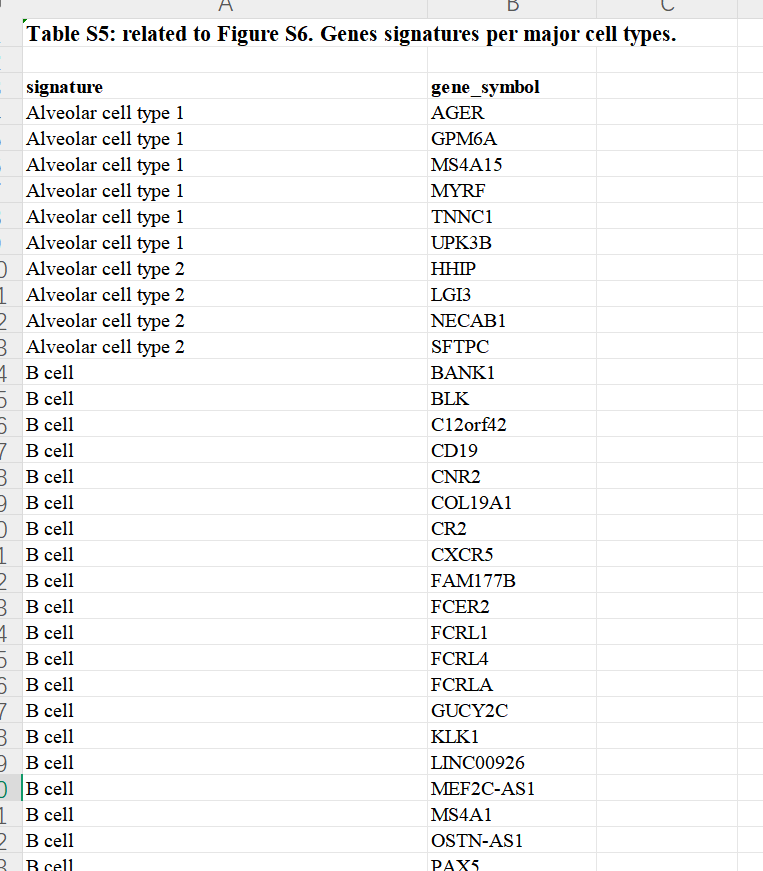
还有注释的marker表格:
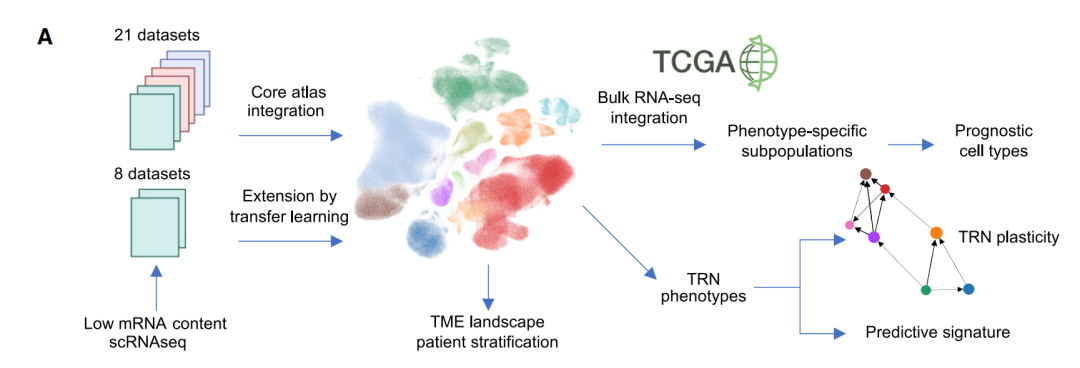
文章分析大纲
通过整合19项研究、21个数据集共计298名患者的505份样本(图1A),「首次构建了非小细胞肺癌核心图谱」。这一综合性单细胞图谱汇集了经专家审定、质量保证且经过预分析的转录组数据,数据来源包括公开研究及我们自有数据集(UKIM-V),涵盖所有组织学类型的早期与晚期非小细胞肺癌。
非小细胞肺癌核心图谱
该核心图谱共整合898,422个单细胞,基于既往建立的经典单细胞标志(图S1A)将其注释为12个粗粒度细胞类型及44个主要细胞亚型/状态(如分裂期细胞),包括169,223个上皮细胞、670,409个免疫细胞及58,790个基质与内皮细胞(图1B)。通过已报道的标志基因22(图S1J),我们进一步标注了关键CD8+ T细胞亚群(终末耗竭型、活化型、效应记忆型、初始型、NK样、分裂期)。

细胞间通讯分析
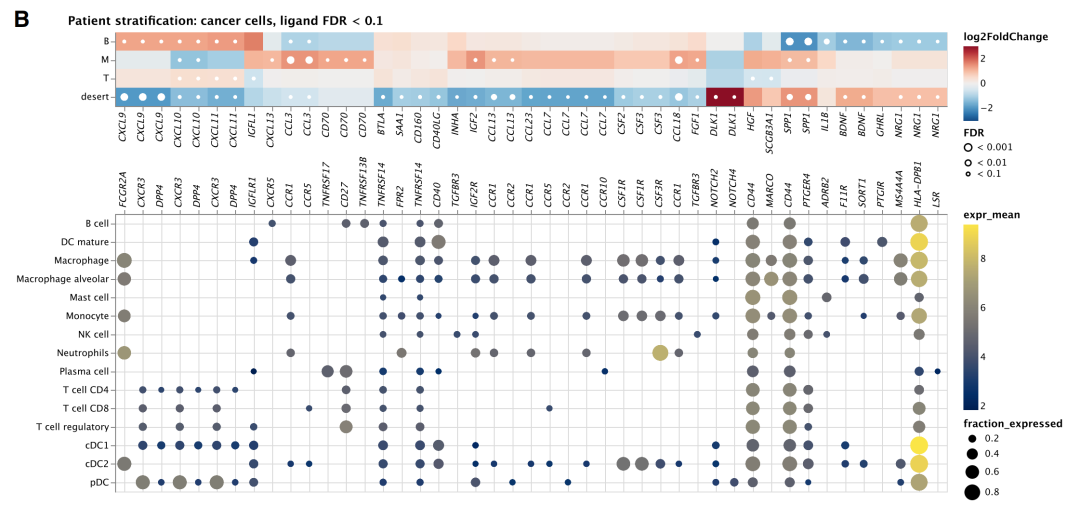
LUAD与LUSC中癌细胞向主要免疫细胞进行通讯的Circos图。展示前10个差异表达的癌细胞配体。红色连线表示在LUAD中上调的相互作用,蓝色连线表示在LUSC中上调的相互作用。

各患者亚型中的癌-免疫细胞通讯。上图:各亚型(B、M、T、ID)中癌细胞的差异表达配体(基于伪批量数据的DESeq2分析,FDR < 0.1)。下图:相应受体及其按细胞类型的表达情况。点的大小和颜色分别表示表达受体的细胞比例和基因表达水平(所有患者的平均值)。仅显示在相应细胞类型中至少10%的细胞表达的受体数据点。
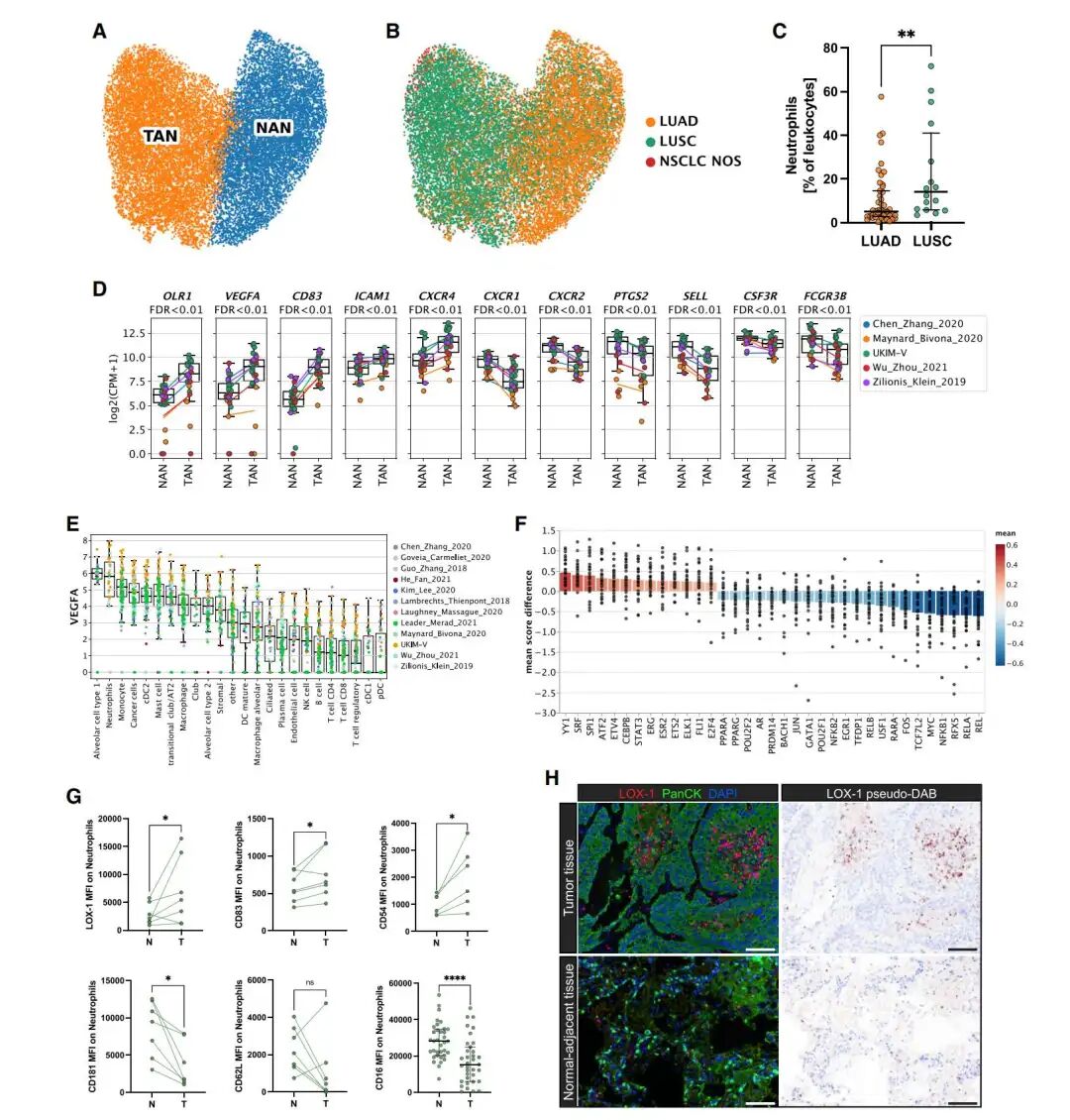
组织驻留中性粒细胞特征
A-B:扩展图谱中组织驻留中性粒细胞(TRNs)的UMAP可视化:(A)按肿瘤相关中性粒细胞(TANs)与癌旁组织中性粒细胞(NANs)分类;(B)按组织病理学类型着色(依据组织病理学评估定义)。
C:流式细胞术检测LUAD与LUSC肿瘤组织中中性粒细胞比例(占白细胞百分比)(LUAD n=47,LUSC n=16;Wilcoxon检验,**p<0.01)。水平线代表中位数,须线表示四分位距。
D:TAN候选基因筛选。每个数据点代表同时在NAN和TAN组中至少检出10个中性粒细胞的患者。连线表示各研究的均值。p值通过配对t检验计算并经FDR校正。
E:原发肿瘤样本中不同细胞类型VEGFA表达水平。每个数据点代表至少检出10个细胞的患者(中位数展示,箱线图表示四分位距,须线范围为1.5倍IQR)。
F:基于DoRothEA的TAN与NAN转录因子分析。每个数据点代表单个患者,条形表示所有患者均值。展示平均评分差异>0.2且FDR<0.1的转录因子,p值经配对t检验与FDR校正。
G:流式细胞术验证选定候选基因在肿瘤与癌旁样本中的差异。每个数据点代表未纳入scRNA-seq数据集的病例。配对Wilcoxon检验,p<0.05,***p<0.0001。CD16图示:水平线代表中位数,须线表示四分位距。
H:LUSC患者肿瘤组织与匹配癌旁肺组织中LOX-1(红色)与全细胞角蛋白(绿色)的多重免疫荧光染色。比例尺:100微米。
今天分享到这~
如果上面的内容对你有用,欢迎一键三连!
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-10-28,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

