R中CSV与CSV的比较
R中CSV与CSV的比较
提问于 2018-11-15 23:29:08
我需要比较R中的两个csv文件,并编写两个文件中不匹配的记录。我能够用下面的代码完成上面的任务,
library(dplyr)
a <- c("ads", "ads", "abc")
b <- c(121, 345, 23.300)
c <- c(21,22,23)
srce <- cbind.data.frame(a,b,c)
d <- c("ads", "ds", "abc")
e <- c(121, 345, 23)
f <- c(21,22,23)
trgt <- cbind.data.frame(d, e, f)
colnames(trgt) <- colnames(srce)
#Compare csv files
nn <- anti_join(srce, trgt)最后的输出给了我不匹配的行,
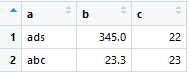
但我需要找出两个文件中不匹配的细胞,
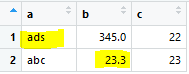
是否有办法识别不匹配的细胞,而不是整个记录?
谢谢Balaji.SJ
回答 1
Stack Overflow用户
回答已采纳
发布于 2018-11-15 23:41:11
如果您将stringsAsFactors = FALSE参数与cbind.data.frame一起使用,那么一个简单的逻辑比较就可以了:
library(dplyr)
a <- c("ads", "ads", "abc")
b <- c(121, 345, 23.300)
c <- c(21,22,23)
srce <- cbind.data.frame(a,b,c, stringsAsFactors = FALSE)
d <- c("ads", "ds", "abc")
e <- c(121, 345, 23)
f <- c(21,22,23)
trgt <- cbind.data.frame(d, e, f, stringsAsFactors = FALSE)
colnames(trgt) <- colnames(srce)
# logical comparison:
srce == trgt
a b c
[1,] TRUE TRUE TRUE
[2,] FALSE TRUE TRUE
[3,] TRUE FALSE TRUE若要比较两个数据并提取不匹配列的列名,请计算矩阵colSums的srce != trgt,并使用colSums > 0显示所有列的名称。
names(which(colSums(srce != trgt, na.rm = TRUE) > 0))
[1] "a" "b"页面原文内容由Stack Overflow提供。腾讯云小微IT领域专用引擎提供翻译支持
原文链接:
https://stackoverflow.com/questions/53333259
复制相关文章
相似问题

![Eclipse中使用SVN Eclipse配置SVN[通俗易懂]](https://ask.qcloudimg.com/http-save/yehe-8223537/caa2e40eabd279997ade5a27c8050ade.png)









