Nat. Methods | Novae: 面向空间转录组的图结构基础模型
Nat. Methods | Novae: 面向空间转录组的图结构基础模型

DrugAI
发布于 2026-01-06 14:01:43
发布于 2026-01-06 14:01:43
DRUGONE
空间转录组技术通过保留组织空间结构,提供细胞级基因表达的高分辨率视图。研究人员提出 Novae——一个能够在多张切片、不同组织、不同基因面板及不同空间组学技术间执行零样本推理的图结构基础模型。Novae 利用近 3,000 万细胞构成的训练数据,学习细胞在组织环境中的空间表示。该模型不仅能够识别空间结构域,还能原生纠正批次效应,并生成嵌套的多尺度空间域结构。此外,Novae 支持空间变异基因分析、空间通路分析及空间轨迹推断,为空间转录组学研究提供稳健且可迁移的基础架构。

空间转录组数据揭示细胞与其微环境之间的空间关系,是研究组织功能与疾病进展的重要工具。与传统 scRNA-seq 相比,空间转录组保留空间定位,从而更易识别微环境结构域。但随着技术快速发展,实验往往使用不同机器或不同基因面板,导致显著的批次效应,使跨切片分析困难。
传统方法依赖固定基因集、外部批次校正工具,并在多切片比较上表现有限。同时,不同空间平台(NGS vs Imaging-based)在分辨率与基因覆盖范围上差异巨大,使跨面板学习更加挑战。研究人员因此需要一个能够原生整合多技术、跨组织且可泛化的基础模型。
方法
研究人员构建 Novae——一个基于图注意力网络的自监督基础模型。模型将细胞视为图节点,并以邻域空间关系作为图结构。通过对近 3,000 万细胞、来自 18 种组织与多平台(Xenium、MERSCOPE、CosMX)的数据进行训练,Novae 能学习细胞局部环境的空间表示。模型包含:空间邻域编码、自监督对比学习、跨面板学习策略、原生批次效应校正,以及层级空间结构域生成机制。Novae 可直接用于零样本推理,也可根据任务执行微调。
结果
Novae 作为空间转录组基础模型的整体能力
- 能在不重新训练的情况下执行零样本空间域预测;
- 可选择细粒度分辨率,生成嵌套式层级空间域;
- 原生进行批次效应校正,使跨切片的空间域可比较;
- 在任意基因 panel、组织类型与平台上均可使用。
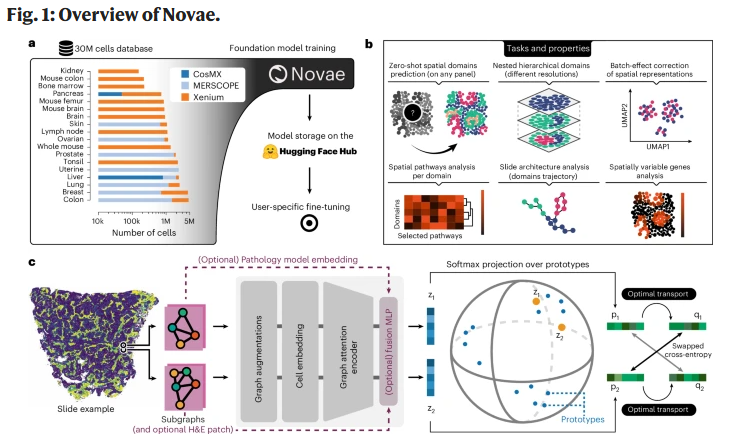
图1:Novae 训练数据、模型结构、零样本推理
多切片、多 panel 条件下的空间域一致性与连续性提升
研究人员在三种测试场景中,将 Novae 与 SpaceFlow、GraphST、SEDR、STAGATE、NicheCompass 等方法对比:
(1) 乳腺组织(跨不同基因面板)
- Novae 使用所有 panel 联合训练;
- 其他模型只能使用交集的 185 个基因;
- Novae 在 FIDE(域边连续性)与 JSD(跨切片一致性)上均显著优于基线;
- 即使在零样本模式下,模型仍保持高性能。
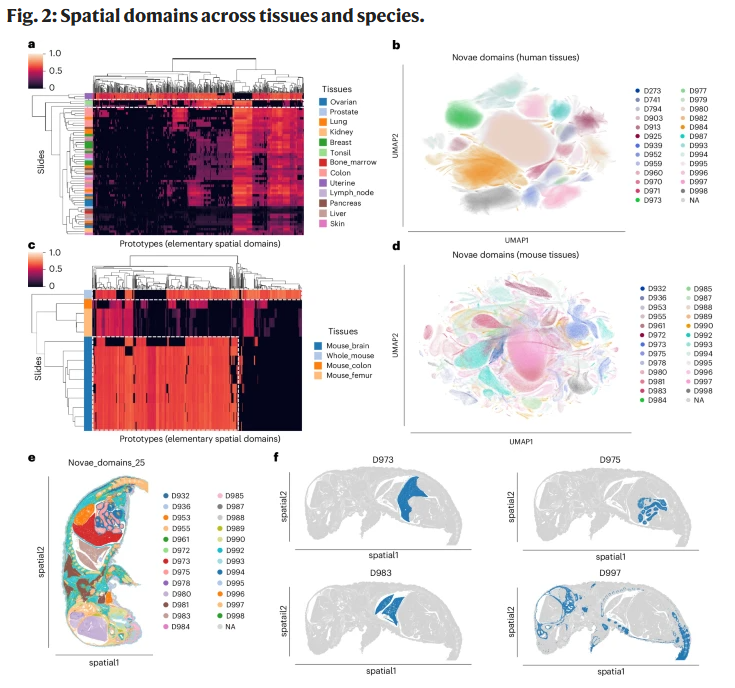
图2:乳腺组织跨 panel 空间域评估
(2) 结肠组织(基因交集极小)
- Novae 可跨所有切片训练;
- 其他模型需为每个切片单独训练并合并表示;
- Novae 在两种模式(zero-shot & fine-tuned)中均取得最佳表现。
(3) 空间可视化与生物学解释性
- Novae 更好识别跨切片共享的 stromal、glandular 等空间域;
- 能区分免疫细胞聚集区域与肿瘤克隆扩张区域;
- 批次校正后,其潜在空间显示更高一致性,但不会过度校正。
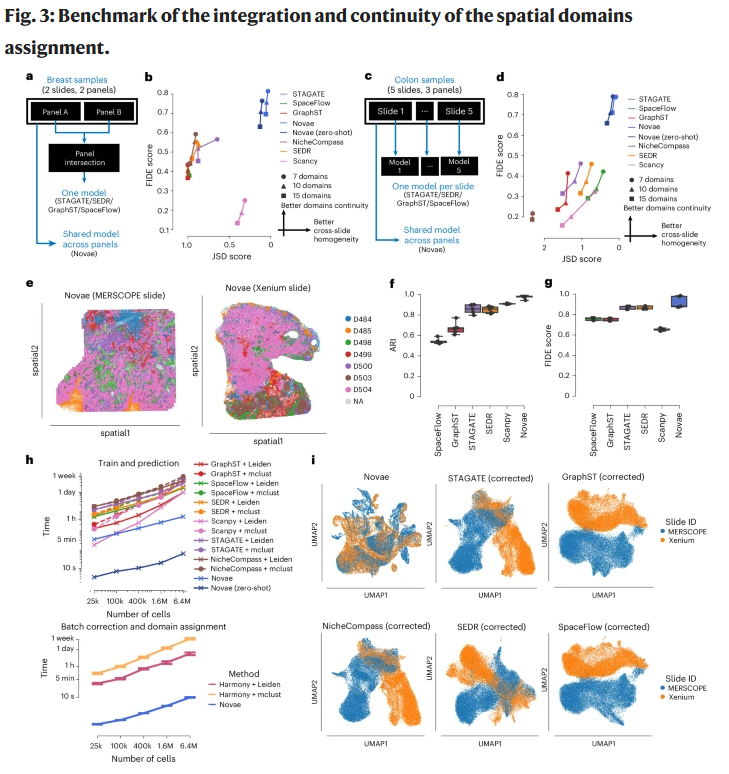
图3:结肠组织跨切片空间域评估
嵌套空间域结构与跨分辨率稳定性
Novae 提供多层级空间域,从粗粒度组织区块到细粒度亚结构域,能够连续追踪组织空间层次结构,克服单尺度模型的局限。
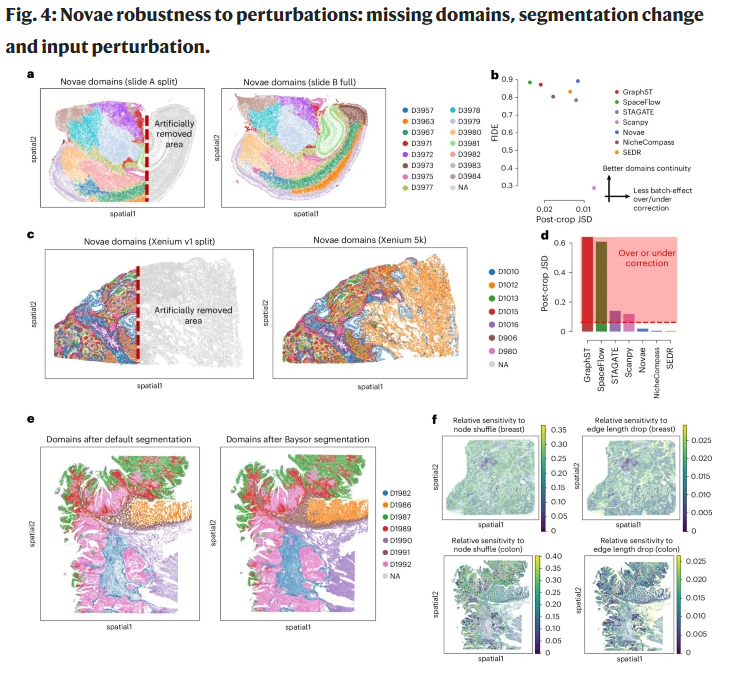
图4:嵌套空间域的多尺度表示
下游任务的广泛适用性
Novae 的空间表示可用于多种任务:
- 空间轨迹(domain trajectory)分析;
- 空间变异基因(SVG)分析;
- 空间通路富集分析;
- 切片结构与组织构型研究。
这些任务无需重复运行外部工具,并能在改变空间域分辨率时保持一致性。
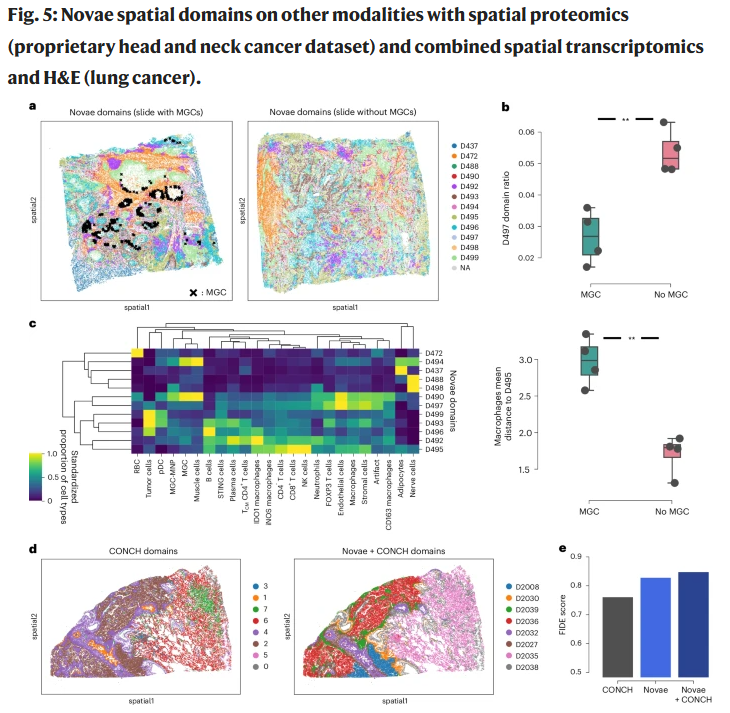
图5:Novae 在下游任务中的应用
批次效应的原生纠正与跨平台泛化
不同技术(如 Xenium vs MERSCOPE)常存在显著批次差异。Novae 通过图结构与训练策略原生调整这些差异,可在无需额外工具(如 Harmony)的条件下实现统一的空间结构域识别。
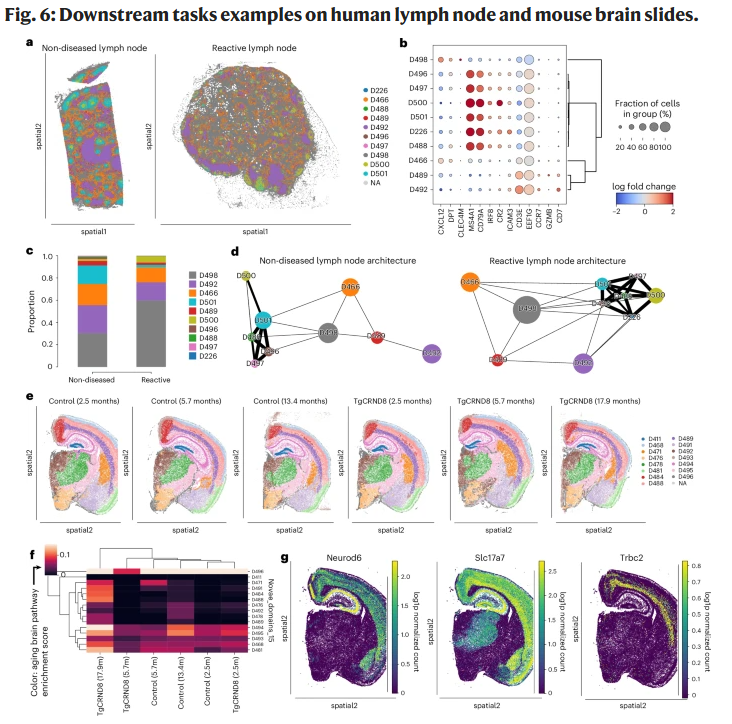
图6:原生批次效应校正与潜在空间对齐效果
讨论
Novae 作为空间转录组的图结构基础模型,首次实现了:
- 跨组织、跨平台、跨基因 panel 的大规模统一建模;
- 无需 retraining 的零样本空间域预测;
- 多尺度嵌套空间结构域的构建;
- 原生批次效应校正,避免手工管线复杂性;
- 用于 SVG、路径分析、轨迹分析等多下游任务的通用表示。
与传统方法相比,Novae 消除了对固定基因集、外部批次校正与额外聚类工具的依赖,大大提升了空间转录组分析的稳定性与可推广性。研究人员预计该模型将成为未来空间组学工具链中的关键基础模块,特别适用于多切片研究、跨实验整合与组织结构域解析。
整理 | DrugOne团队
参考资料
Blampey, Q., Benkirane, H., Bercovici, N. et al. Novae: a graph-based foundation model for spatial transcriptomics data. Nat Methods 22, 2539–2550 (2025).
https://doi.org/10.1038/s41592-025-02899-6
内容为【DrugOne】公众号原创|转载请注明来源
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-12-11,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读

