Briefings Bioinf. | GLDM:基因表达谱引导的图潜在扩散模型实现100%有效分子生成
Briefings Bioinf. | GLDM:基因表达谱引导的图潜在扩散模型实现100%有效分子生成

MindDance
发布于 2026-01-22 12:31:48
发布于 2026-01-22 12:31:48
定向发现具有理想生物活性的先导化合物是计算机辅助药物设计的核心挑战。现有方法多基于 SMILES 表示,在基因表达谱条件生成中化学有效性低于10%。新加坡南洋理工大学团队在《Briefings in Bioinformatics》发表的研究提出了 GLDM(图潜在扩散模型)——一个在潜空间操作的扩散模型,通过基于主题的图生成和交叉注意力机制,实现了100%化学有效性的分子生成。在 GuacaMol 基准测试中,GLDM 全面超越现有方法,并在 EGFR、HDAC 等9个重要靶点的分子对接实验中展现出优异的结合亲和力。

1. 药物发现的双重挑战
1.1 化学有效性与生物活性的权衡
传统的药物发现流程需要筛选数百万甚至数十亿个候选分子,成本高昂且耗时漫长。深度生成模型的出现为这一领域带来了新希望,但现有方法面临两大核心挑战:
化学有效性不足:大多数现有的深度学习方法基于 SMILES(简化分子线性输入规范)表示,通过字符串生成分子。这种方式虽然简单,但生成的分子往往违反化学规则(如价键错误、环结构不合理等)。研究表明,现有基于 SMILES 的基因表达谱条件生成方法,化学有效性低于10%。
生物活性导向缺失:大多数生成模型关注化学性质(如分子量、溶解度、合成可及性),但很少考虑生物学背景。理想的药物分子不仅需要化学有效,还需要能够与特定的生物靶点结合,产生期望的生物学效应(如抑制癌症相关蛋白、调控基因表达等)。
1.2 GLDM 的核心突破
为解决这两大挑战,南洋理工大学团队开发了 GLDM,实现了三大突破:
- 100%化学有效性:通过图表示和基于主题的生成策略,确保所有生成分子完全符合化学规则
- 生物活性引导:利用基因表达谱作为条件,通过交叉注意力机制定向生成具有特定生物活性的分子
- 潜空间高效性:在低维潜空间而非高维原始空间操作,显著提高了生成效率和质量
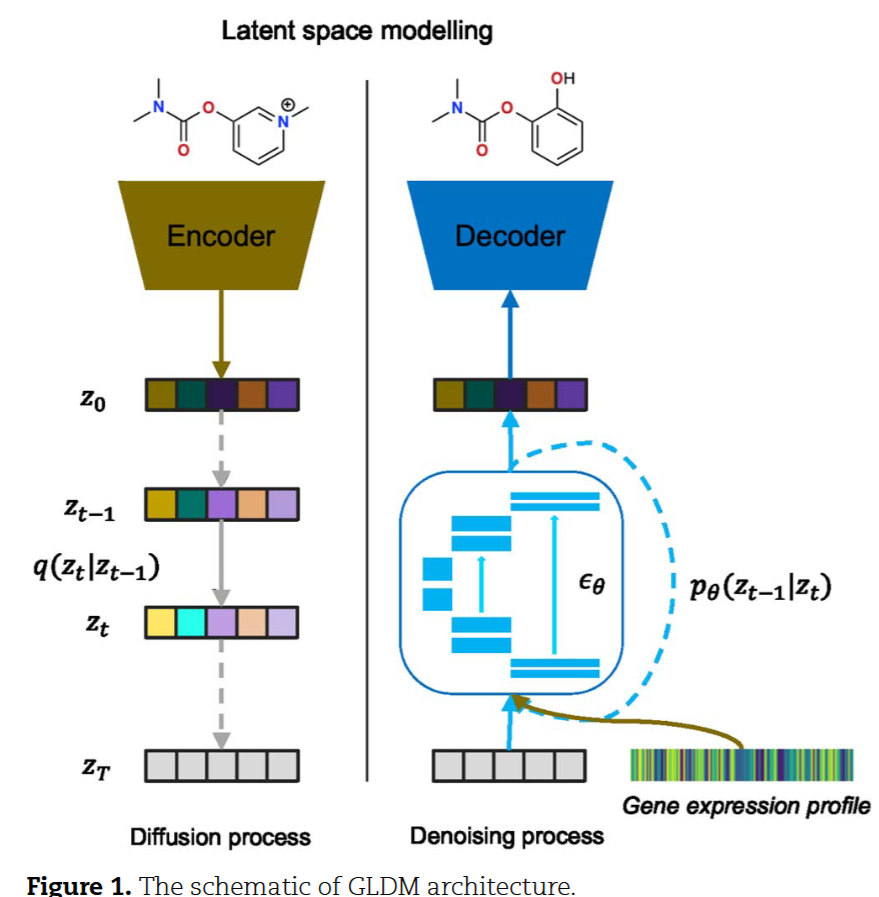
GLDM整体架构示意图,展示自动编码器、潜空间扩散过程和条件生成流程
GLDM整体架构示意图,展示自动编码器、潜空间扩散过程和条件生成流程
2. 核心技术创新
2.1 图表示:从 SMILES 到分子图
与基于 SMILES 的字符串生成不同,GLDM 采用分子图表示:将分子表示为图结构,其中节点代表原子,边代表化学键。这种表示方式的优势在于:
- 显式编码化学规则:通过图的连接关系直接表达化学键的拓扑结构,避免了 SMILES 中隐式的语法错误
- 保留空间信息:图结构能够更好地捕捉分子的三维空间特征,这对于理解分子与蛋白质的相互作用至关重要
- 支持价键检查:在生成过程中可以实时检查每个原子的价态是否合理,确保化学有效性
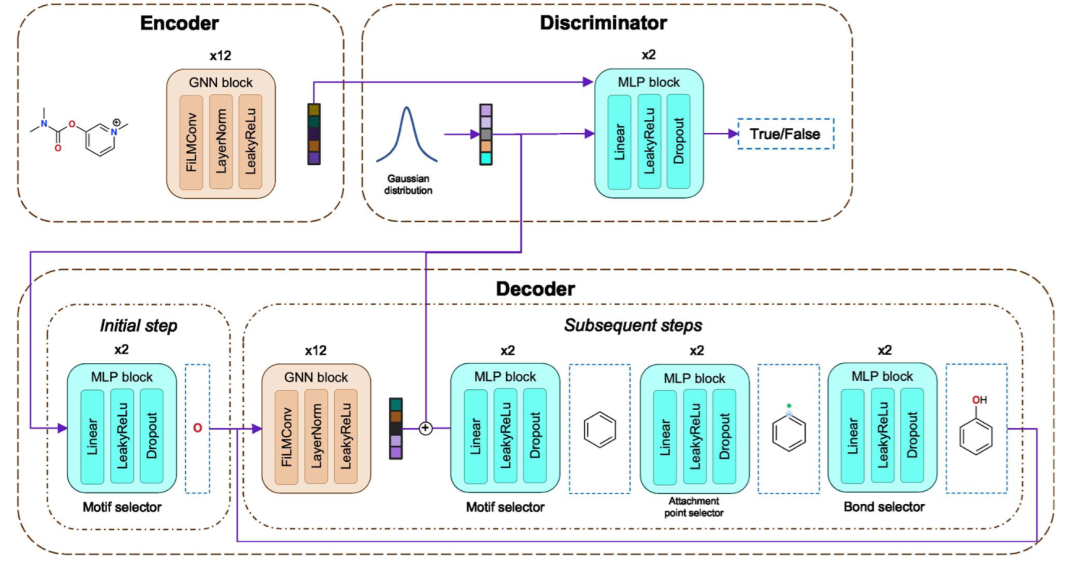
自动编码器架构图,展示基于图神经网络(GNN)的编码器和基于主题选择的解码器
自动编码器架构图,展示基于图神经网络(GNN)的编码器和基于主题选择的解码器
2.2 潜空间扩散:压缩与生成的结合
GLDM 的核心架构包含三个关键组件:
图自动编码器:
- 编码器:使用图神经网络(GNN)将复杂的分子图压缩为低维潜向量
- 解码器:采用基于主题的生成策略,将潜向量重建为分子图
潜空间扩散模型:在潜向量空间进行扩散过程,而非在原始的高维分子图空间。这带来两大优势:
- 计算效率:潜空间维度远低于原始图空间,加速了扩散过程
- 生成质量:潜空间的平滑性使得扩散模型更容易学习数据分布
2.3 基于主题的生成:从原子到分子片段
传统的图生成模型逐个原子地构建分子,这种方式容易产生化学上不合理的中间状态。GLDM 引入了基于主题的生成策略:
主题是指化学上有意义的分子片段(如苯环、羧基、氨基等)。GLDM 的解码器包含一个主题选择器,它:
- 从预定义的主题库中选择合适的化学片段
- 将这些片段组装成完整的分子
- 确保片段之间的连接符合化学规则
这种策略大幅提高了生成分子的化学有效性和合理性。
2.4 交叉注意力机制:基因表达谱引导生成
为了实现生物活性导向的分子生成,GLDM 在扩散模型的去噪网络中引入了多头交叉注意力机制:
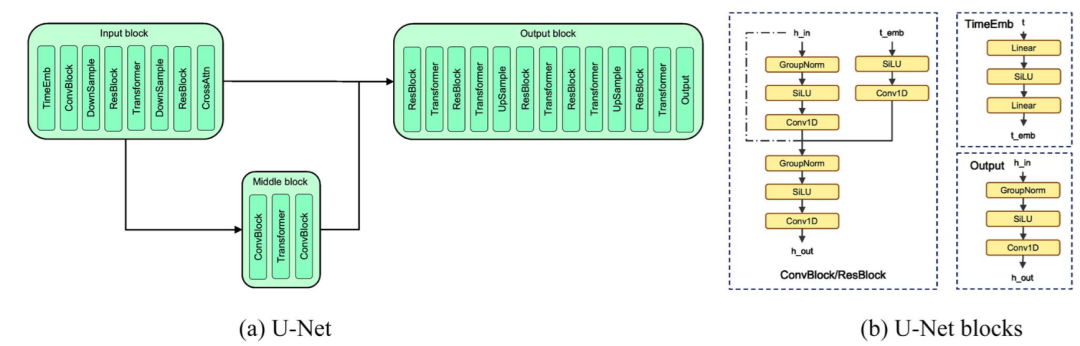
U-Net 骨干网络架构,展示交叉注意力层如何将基因表达谱作为条件信号整合到去噪过程中
U-Net 骨干网络架构,展示交叉注意力层如何将基因表达谱作为条件信号整合到去噪过程中
基因表达谱作为条件:基因表达谱描述了某种生物学状态下(如疾病状态、药物处理后)各个基因的表达水平。GLDM 将基因表达谱编码为条件向量。
交叉注意力融合:在扩散模型的每一步去噪过程中,交叉注意力机制让模型能够"关注"基因表达谱中的关键信息,从而生成与特定生物学背景相匹配的分子。
例如,如果基因表达谱显示某个癌症相关基因高表达,模型会倾向于生成能够抑制该基因或其蛋白产物的分子。
3. 性能验证与关键发现
3.1 GuacaMol 基准测试:全面超越现有方法
GuacaMol 是分子生成领域最权威的基准测试之一,包含约150万个分子。GLDM 在多个关键指标上取得了出色表现(详见 Table 2):
化学有效性:
- GLDM:1.0(100%)
- DiGress(图扩散基线):0.852
- 其他 SMILES 方法:< 0.10(在条件生成场景下)
独特性与新颖性:
- GLDM Uniqueness:0.999
- GLDM Novelty:0.997
这表明 GLDM 生成的几乎每个分子都是唯一且新颖的,避免了简单的记忆训练数据。
分布学习指标:
- KL Divergence:0.926(越接近1越好,表示生成分布与真实分布接近)
- Fréchet ChemNet Distance (FCD) :0.424(越低越好,DiGress 为0.680)
FCD 衡量生成分子与真实分子在化学特征空间中的距离。GLDM 的低 FCD 值表明其生成的分子在化学性质上更接近真实药物分子。
3.2 约束生成:平衡合成可及性与药物相似性
除了无约束生成,GLDM 还在约束生成任务中表现出色。研究团队评估了生成分子的两个关键药物化学性质:
合成可及性(Synthetic Accessibility, SA):评分越低表示分子越容易合成(1-10分制)药物相似性(Quantitative Estimate of Drug-likeness, QED):评分越高表示分子越符合类药性质(0-1分制)
与基线方法 BiAAE 相比,GLDM 在这两个指标上均表现更优。特别是在使用 VAE 损失训练的版本中,GLDM 生成分子的 QED 得分甚至高于原始数据集,展示了其生成高质量候选药物的能力。
3.3 消融实验:正则化损失的影响
研究团队系统地比较了三种正则化损失函数对自动编码器的影响:VAE、GAN 和 WAE(详见消融实验结果)。
GAN 损失的双刃剑:
- 优势:在 KL Divergence(0.926)和 FCD(0.424)上表现最好,说明生成的分子分布最接近真实数据
- 劣势:生成的分子往往过于复杂(如包含大环、长链结构),导致合成可及性(SA)变差
WAE 损失的平衡方案:
- 能够在保持良好结合亲和力的同时,兼顾 SA 和 QED 分数
- 被认为是发现可合成、类药且具有生物活性的先导化合物的最佳选择
这一发现对实际药物发现具有重要指导意义:并非所有在分布学习指标上表现最好的模型都能生成实用的药物分子。
3.4 靶点导向生成:9个重要蛋白的实验验证
研究团队针对9个重要的疾病相关蛋白靶点进行了条件生成实验:
- AKT1, AKT2(PI3K/AKT 信号通路,癌症)
- EGFR(表皮生长因子受体,癌症)
- AURKB(极光激酶B,细胞分裂)
- HDAC1(组蛋白去乙酰化酶,表观遗传调控)
- SMAD(TGF-β 信号通路)
- MTOR(雷帕霉素靶蛋白,细胞生长)
- PIK3CA(磷脂酰肌醇-3-激酶,癌症)
- TP53(肿瘤抑制因子p53)
分子对接验证:研究团队从 PDB 数据库中选择了10个结合姿态结构(详见 Table 4),使用 AutoDock Vina 对生成的分子进行对接评分。
关键结果(以 EGFR 为例):
- PDB ID: 4HJO(EGFR 与已知抑制剂复合物)
- GLDM 生成的分子中,有 73.8% 展现出高结合亲和力(Vina 得分优于参考配体)
- 对比 BiAAE 基线:GLDM 在多个 PDB 结构(如 1M17, 6HSK)上的 Vina 得分均显著更优
HDAC 家族靶点:
- GLDM 在 HDAC8 和 HDAC2 靶点上也展现出优异表现
- 生成的分子不仅结合亲和力强,且结构多样性高,为后续的先导化合物优化提供了丰富的起点
4. 实际应用价值与启示
4.1 从数据驱动到机制驱动
GLDM 的一个重要贡献是展示了如何将生物学机制(通过基因表达谱体现)整合到生成模型中。传统的分子生成模型大多是"盲目"的——它们生成化学上合理的分子,但不考虑这些分子是否具有期望的生物学功能。
GLDM 的条件生成机制使得:
- 研究人员可以根据疾病的基因表达特征"定制"分子
- 模型能够学习基因表达模式与分子结构之间的隐含关系
- 生成的分子更有可能在实际生物学实验中展现出活性
4.2 100%化学有效性的意义
在药物发现的早期阶段,化学有效性是硬性要求。如果生成的分子违反化学规则,即使在计算机上"看起来"有前景,也无法在实验室中合成和测试。
GLDM 实现的100%化学有效性意味着:
- 所有生成的分子都可以直接交给合成化学家进行实验验证
- 节省了人工筛选和修正无效分子的时间
- 提高了从计算预测到实验验证的转化效率
4.3 基于主题生成的优势
基于主题的生成策略体现了领域知识与深度学习的结合:
- 药物化学知识:许多成功的药物都包含特定的药效团(pharmacophore),如苯环、杂环等
- 生成效率:逐个原子生成需要数百步,而基于主题生成可能只需数十步
- 结构合理性:主题本身就是化学上稳定的片段,降低了生成不合理结构的风险
5. 局限性与未来方向
尽管 GLDM 取得了显著成果,研究团队也坦诚地指出了当前的局限性:
5.1 复杂性与可合成性的权衡
研究发现,GAN 损失虽然在分布学习指标上表现最好,但会导致生成过于复杂的分子(如大环、长链结构)。这些分子的合成难度大大增加,在实际药物开发中可能不切实际。
未来方向:
- 引入合成可及性预测器作为显式约束,在生成过程中实时评估分子的合成难度
- 探索多目标优化策略,在生物活性、化学有效性和合成可及性之间找到最优平衡
5.2 三维构象的考虑
当前的 GLDM 主要在二维分子图层面操作,未充分考虑分子的三维构象信息。然而,分子与蛋白质的结合高度依赖于三维空间结构。
未来方向:
- 整合三维分子生成模型(如最近的 3D 扩散模型)
- 结合构象采样技术,为每个生成的分子生成多个可能的三维构象
- 与 AlphaFold3、Boltz 等结构预测工具联用,预测分子-蛋白质复合物结构
5.3 实验验证的闭环
虽然 GLDM 在计算层面表现出色,但最终的药物发现仍需要实验验证。当前的工作流是单向的:模型生成 → 实验验证。
未来方向:
- 建立主动学习框架:将实验反馈整合回模型训练,形成闭环
- 与自动化实验室(如前文介绍的自主实验室)结合,加速从生成到验证的周期
- 利用小样本学习技术,从有限的实验数据中快速迭代改进模型
5.4 更广泛的生物学条件
当前的条件生成主要基于基因表达谱,但药物发现中还有其他重要的生物学信息:
- 蛋白质结构信息:结合口袋的三维结构、关键残基信息
- 药代动力学性质:ADMET(吸收、分布、代谢、排泄、毒性)
- 多靶点效应:许多疾病需要多靶点药物(如抗癌药)
未来方向:
- 开发多模态条件生成模型,整合序列、结构、表达谱等多种生物学信息
- 探索分层条件机制:在不同的生成阶段引入不同类型的条件
6. 对药物发现的启示
GLDM 的成功展示了几个重要的趋势:
图表示的优势:相比 SMILES,图表示在确保化学有效性和捕捉分子拓扑特征方面具有显著优势。这一趋势也体现在其他最新的研究中(如前文介绍的 DrugCLIP、GCLDM 等)。
潜空间方法的普及:在潜空间而非原始空间操作已成为分子生成的主流范式(如 LDMol、COATI-LDM 等)。这不仅提高了计算效率,也使得模型能够学习到更抽象、更高层次的化学特征。
生物学导向的必要性:纯粹基于化学规则的生成已不足够,将生物学背景(基因表达、蛋白质结构、疾病机制)整合到生成过程中,是实现真正有用的药物发现AI的关键。
开放科学的重要性:虽然本文未明确提及开源,但在药物发现AI领域,开放数据、开放模型、开放验证的趋势日益明显(如前文的 Boltz、DrugCLIP)。GLDM 使用的 GuacaMol、LINCS L1000 等都是公开数据集,为社区提供了可复现的基准。
值得思考的问题
Q1:为什么基于 SMILES 的方法化学有效性如此低? SMILES 是一种字符串表示,本质上是一种语言。生成 SMILES 类似于生成句子——模型需要学习复杂的语法规则。即使是微小的语法错误(如括号不匹配、芳香性标记错误),也会导致整个分子无效。相比之下,图表示直接编码了分子的拓扑结构,且可以在生成每一步进行价键检查,大大降低了产生无效分子的风险。此外,SMILES 的同一个分子可能有多种不同的字符串表示,增加了学习难度。
Q2:GLDM 的100%有效性是否意味着牺牲了多样性? 这是一个很好的问题。从结果来看,GLDM 在保持100%有效性的同时,Uniqueness(0.999)和 Novelty(0.997)也都接近完美,说明多样性并未受到明显影响。这得益于基于主题的生成策略:主题库中包含了多样化的化学片段,模型学习的是如何组合这些片段,而不是简单地记忆训练数据。此外,扩散模型本身的随机性也有助于生成多样化的分子。
Q3:73.8%的高亲和力分子比例在实际药物发现中意味着什么? 在虚拟筛选中,如果能将高亲和力候选物的比例从1-5%(传统虚拟筛选的典型水平)提升到70%以上,这将极大地提高实验验证的效率。以往可能需要合成和测试数百个化合物才能找到几个有希望的先导物,现在可能只需要合成数十个。这不仅节省了时间和成本,也加速了整个药物发现流程。当然,需要注意的是,计算预测的亲和力与实验测量的亲和力之间仍存在差距,73.8%是基于 Vina 对接得分的预测,最终仍需实验验证。
Q4:交叉注意力机制如何"理解"基因表达谱? 交叉注意力机制本质上是一种特征对齐:它让模型学习潜向量(代表分子)中的哪些部分应该与基因表达谱(代表生物学状态)中的哪些部分相关联。例如,如果基因表达谱显示某个癌症驱动基因高表达,注意力机制可能会学习到:生成具有特定官能团(能够与该基因产物蛋白结合)的分子。这一过程是端到端学习的,模型并不需要显式告知哪些基因对应哪些化学结构,而是从数据中自动学习这种映射关系。
Q5:WAE 损失为何在实际应用中更优? WAE(Wasserstein Autoencoder)损失使用 Wasserstein 距离来衡量潜空间分布与先验分布的差异。相比 VAE 的 KL 散度,Wasserstein 距离能够更好地处理分布不匹配的情况。在药物发现场景下,WAE 损失能够:(1)避免过度正则化,保留更多的分子多样性;(2)在保持生成质量的同时,不会像 GAN 那样产生过于复杂的分子。实验结果显示,WAE 在 SA、QED 和结合亲和力三者之间取得了最佳平衡,这正是实际先导化合物发现所需的。
Q6:GLDM 能否用于多靶点药物设计? 理论上可以。当前的 GLDM 接受单个基因表达谱作为条件,但可以扩展为接受多个基因表达谱或多个靶点结构的联合条件。例如,可以设计一个多条件交叉注意力模块,同时考虑多个靶点的信息。在训练数据方面,需要多靶点活性的分子-基因表达数据对,这在一些公开数据库(如 ChEMBL)中是可获得的。多靶点药物设计是一个重要的方向,尤其是在癌症、神经退行性疾病等复杂疾病的治疗中。
Q7:GLDM 与前文介绍的其他模型(如 LDMol、GCLDM)有何异同? 这些模型都属于潜空间扩散模型家族,但侧重点不同:
- LDMol:使用 SMILES 枚举的对比学习预训练,侧重文本(IUPAC 名称)引导的分子生成
- GCLDM:强调几何完备性,侧重3D分子生成和手性感知
- GLDM:强调基因表达谱条件生成和100%化学有效性
它们的共同点是都利用自动编码器压缩分子表示,再在潜空间进行扩散。不同点在于表示方式(SMILES vs 2D图 vs 3D图)、条件信息(文本 vs 基因表达谱 vs 结构约束)和应用场景(分子优化 vs 从头设计 vs 靶点导向设计)。这些方法是互补的,未来可能会看到将它们的优势结合起来的混合模型。
结语
GLDM 的发布标志着生物学导向的分子生成迈出了坚实的一步。通过整合基因表达谱这一关键的生物学信息,GLDM 不仅实现了100%的化学有效性,更重要的是,它生成的分子具有明确的生物学意义——能够与特定的疾病相关靶点结合。
从技术角度看,GLDM 展示了几个重要的设计原则:图表示优于字符串表示、潜空间操作提高效率、基于主题的生成确保质量、交叉注意力实现条件控制。这些原则在未来的分子生成模型设计中具有重要的参考价值。
从应用角度看,GLDM 在 EGFR、HDAC 等重要癌症靶点上的成功验证,展示了其在实际药物发现中的潜力。73.8%的高亲和力分子比例,意味着实验验证的效率可以大幅提高。当然,从计算预测到临床候选物还有很长的路要走,但 GLDM 为这一旅程提供了一个强有力的起点。
随着技术的不断成熟——整合3D构象、引入合成可及性约束、建立主动学习闭环——我们有理由期待:AI驱动的、生物学导向的、高效的药物发现将成为现实。
参考文献:Conghao Wang, Hiok Hian Ong, Shunsuke Chiba, Jagath C Rajapakse, GLDM: hit molecule generation with constrained graph latent diffusion model, Briefings in Bioinformatics, Volume 25, Issue 3, May 2024, bbae142, https://doi.org/10.1093/bib/bbae142
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2026-01-09,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

