Cell Genomics | 南京大学陈迪俊团队开发一种基于元模块用于单细胞和空间多组学整合与注释通用分析框架
Cell Genomics | 南京大学陈迪俊团队开发一种基于元模块用于单细胞和空间多组学整合与注释通用分析框架

DrugOne
发布于 2026-01-13 15:44:18
发布于 2026-01-13 15:44:18
在当今生命科学前沿,单细胞与空间组学技术正以前所未有的精度描绘生命组织的细胞构成与空间架构。然而,海量多模态、多批次、跨物种数据的整合与解读,一直是阻碍领域发展的核心瓶颈。
近日,南京大学陈迪俊团队在《Cell Genomics》上发表重要研究成果,首创一种名为SSpMosaic的计算框架,首次引入“元模块(metaprograms)”作为生物学通用锚点,打通了单细胞与空间多组学数据的整合、注释与空间解析全流程,为复杂组织与疾病微环境研究提供了强大、统一且可解释的分析工具。
SSpMosaic 的核心是从不同数据集中提取基因共表达或共调控模块,并通过网络传播与层次聚类方法,将其融合为跨样本保守的“元模块”。这些元模块如同细胞状态与功能的“分子指纹”,为后续所有分析任务提供了稳定且可解释的锚点。基于这一基础,SSpMosaic 实现了多模态数据整合、高精度细胞注释、跨分辨率空间解卷积、多组学动态解析,乃至在无单细胞参考情况下的空间结构识别(图1)。
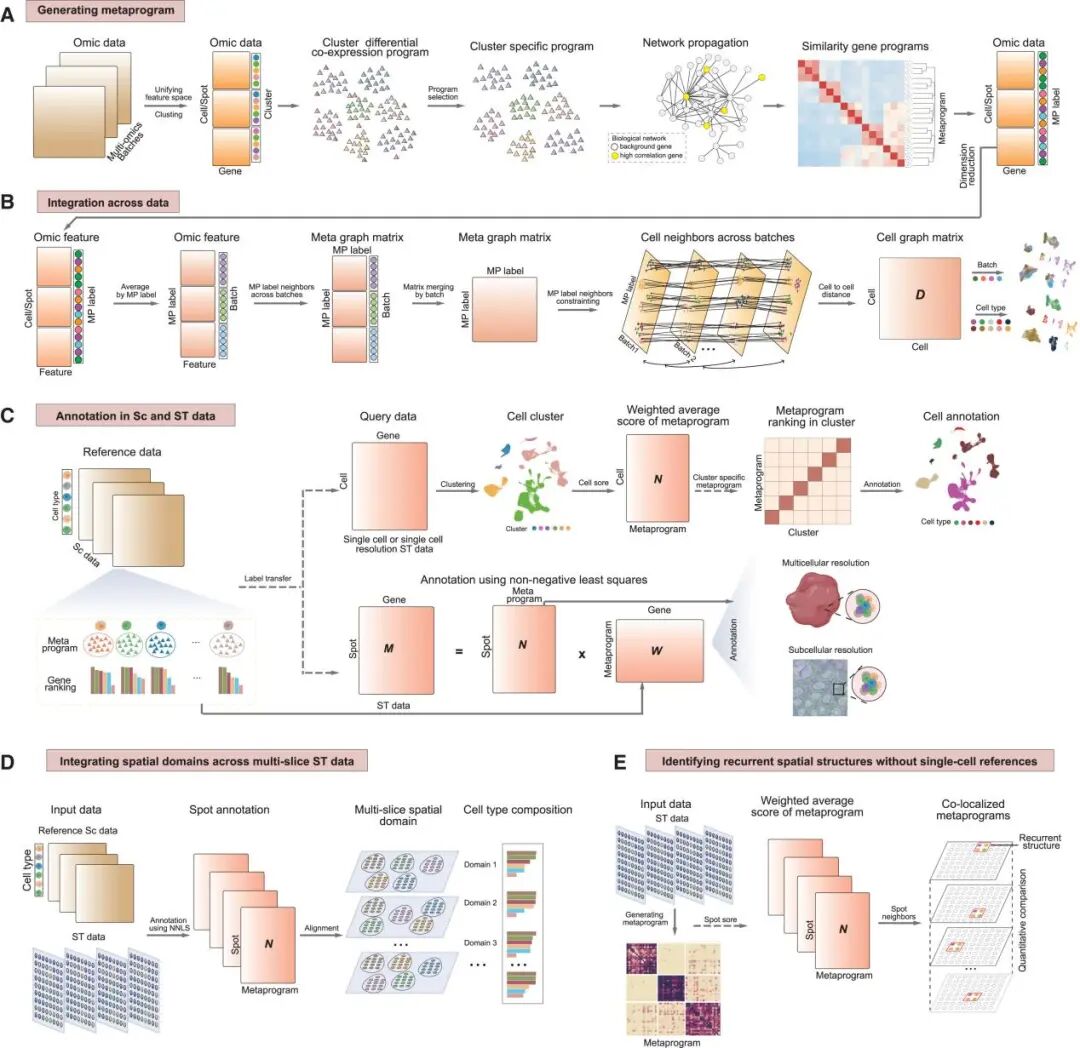
图1. SSpMosaic技术流程图。
在单细胞整合和注释中,SSpMosaic 展现出卓越的性能。它能够无缝整合跨批次、跨物种及跨模态的单细胞数据,例如在结肠癌批次整合、人鼠脑图谱对齐、以及转录组-表观组-蛋白组三模态数据融合中均优于现有方法。在细胞类型注释方面,该框架不仅在大类注释上与专家标注高度一致,还能精细区分高度相似的神经元亚型,并在跨组织数据中自动识别出未被标注的新细胞类群。
针对空间转录组数据,SSpMosaic 实现了从 Visium 点级分辨率到CosMx单细胞分辨率甚至Visium HD的亚细胞分辨率的精准解卷积,准确还原了小鼠嗅球、海马体及人类肺癌组织的空间细胞组成,并进一步识别出具有不同功能特征的空间域。在心肌梗死多组学整合分析中,SSpMosaic 成功解析出 11 个功能空间生态位,动态刻画了疾病进展中的区域特异性变化。
更具创新性的是,SSpMosaic 能够在无配对单细胞数据的情况下,直接对空间转录组进行解析(图2)。在 26 张胶质母细胞瘤切片的分析中,该框架自主推断出 17 个元模块,不仅覆盖已知细胞状态,还识别出如未折叠蛋白反应等新程序。通过新提出的“包裹指数”,研究量化了肿瘤相关巨噬细胞被缺氧区域包裹的空间结构,并揭示了相关信号通路的调控作用。

图2. SSpMosaic实现无单细胞参考的胶质瘤空间微环境解析。
总体而言,SSpMosaic 通过“元模块”这一核心设计,首次建立了一个统一、可解释且扩展性强的分析框架,显著降低了多组学数据整合与空间解析的技术门槛。该工具不仅提升了分析的标准化与可重复性,也为深入理解发育、稳态与疾病中的空间生物学机制提供了强大支撑。
该研究由南京大学生命科学学院博士研究生张月蕾、硕士研究生明文轩为共同第一作者。南京大学生命科学学院陈迪俊副教授、南京大学医学院附属口腔医院邓润智副教授为该论文的共同通讯作者。相关代码已开源,可供学术界免费使用与扩展。
参考资料
Zhang, Yuelei, Wenxuan Ming, Bianjiong Yu, Lele Wang, Kaiyan Lu, Lei Xu, Yanhong Ni, Runzhi Deng, and Dijun Chen. "Robust integration and annotation of single-cell and spatial omics data using interpretable gene programs." Cell Genomics (2025).
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2026-01-13,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读

