听说你的热图聚类乱糟糟
问题
这是一段GEO芯片数据常规的下载、分组、注释、画热图的代码:
rm(list = ls())
library(tinyarray)
a = geo_download("GSE56649")
a$exp = log2(a$exp+1)
library(stringr)
Group= ifelse(str_detect(a$pd$`disease state:ch1`,"control"),"control","patient")
Group = factor(Group,levels = c("control","patient"))
#探针注释
library(hgu133plus2.db)
ids <- toTable(hgu133plus2SYMBOL)
head(ids)
## probe_id symbol
## 1 1007_s_at DDR1
## 2 1053_at RFC2
## 3 117_at HSPA6
## 4 121_at PAX8
## 5 1255_g_at GUCA1A
## 6 1294_at UBA7
exp = trans_array(a$exp,ids)
g = names(tail(sort(apply(exp, 1, sd)),100))
draw_heatmap(exp[g,],Group)
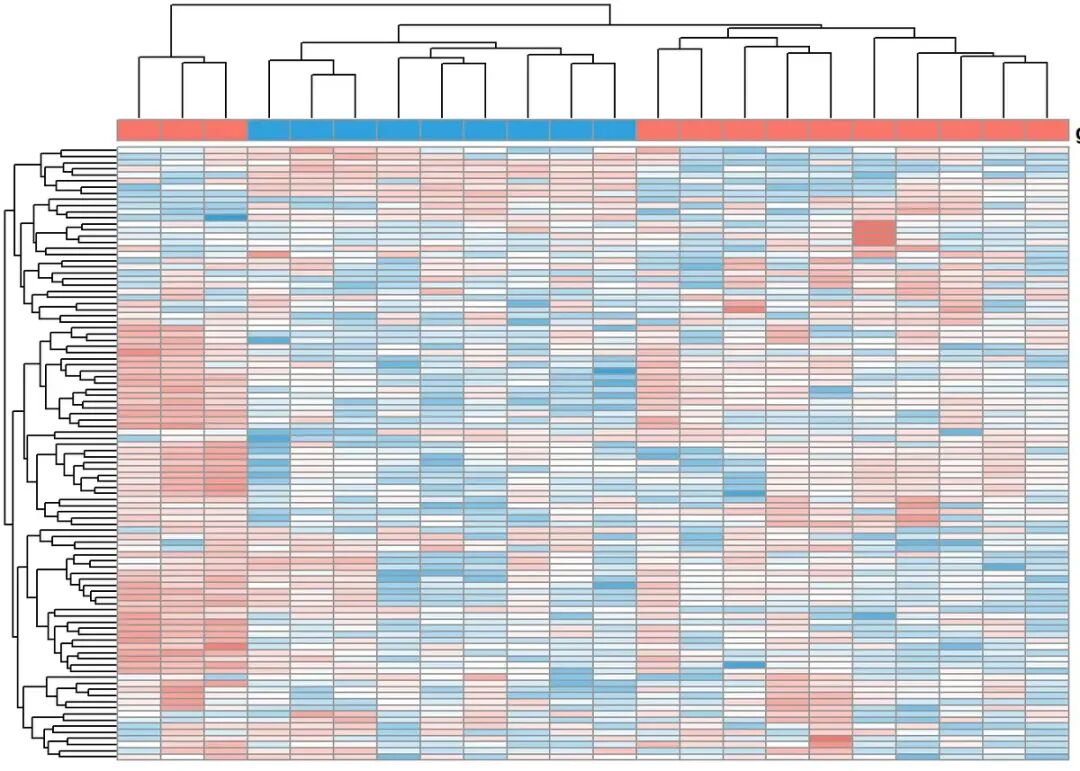
应该是会经常遇到这样的热图,就是说它的聚类情况无法与分组信息吻合。其实这个并不是错了,是因为用于聚类的行(选出的这部分基因)的表达模式在两组之间没有明显区别。
解决办法1:换画图基因
只要改变基因数量,或者换一组基因,聚类树就有可能变得和分组吻合。
解决办法2:取消列的聚类
假如你尝试换基因,还是没有办法让它们变得吻合,那就取消聚类。直接使用热图的参数cluster_cols = F即可。
draw_heatmap(exp[g,],Group,cluster_cols = F)
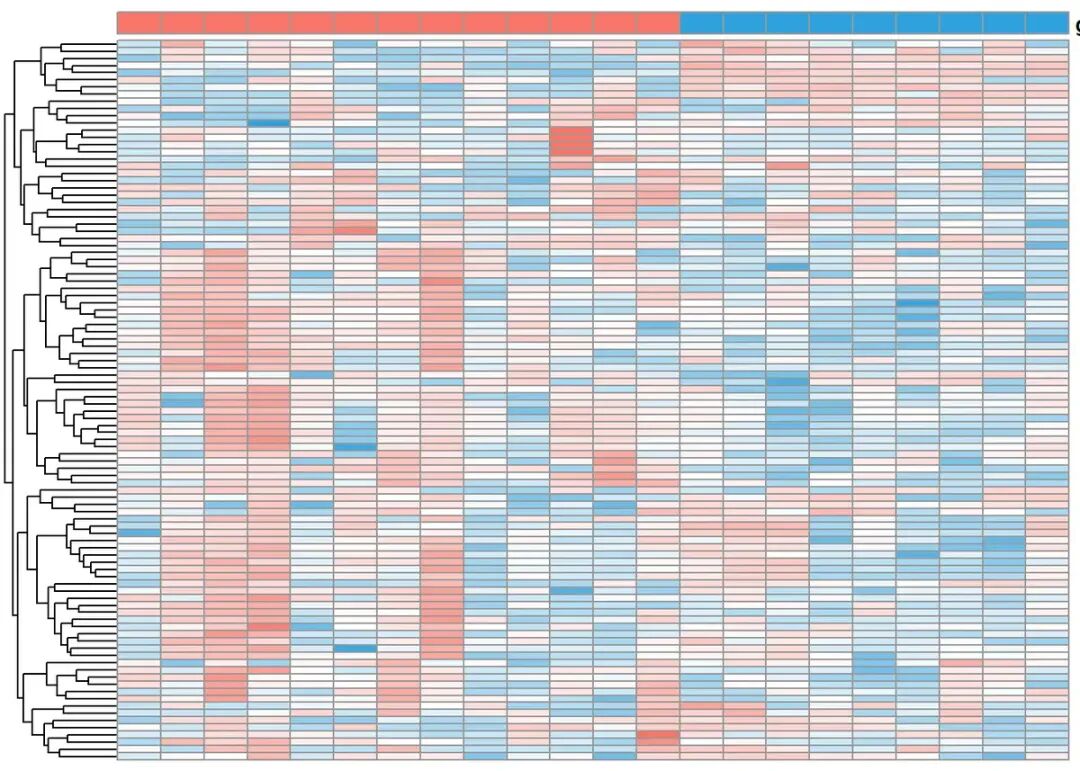
cluster_cols=F控制了列不聚类,热图的列就会按照表达矩阵的列(样本)原本的顺序展示。
解决办法3:排序并取消列聚类
但有的数据它的样本并不是按照分组排好的,而是相间的或者杂乱的(比如control treat treat control control treat这样),仅仅设置cluster_cols=F参数是没有用的。
我们可以手动调整一下表达矩阵的列的顺序,使它按照分组排好,分组信息的顺序也必须跟着调整。
(这个数据的样本本来就是按照分组排好的,调整一下是普适性操作,有则改之无则加勉,排好了再排一次也不影响)
Group
## [1] patient patient patient patient patient patient patient patient patient
## [10] patient patient patient patient control control control control control
## [19] control control control control
## Levels: control patient
anno_col = data.frame(sample = colnames(exp),
Group = Group)
library(dplyr)
anno_col = arrange(anno_col,Group)
exp2 = exp[,anno_col$sample]
Group2 = anno_col$Group
再画图就好了
draw_heatmap(exp2[g,],Group2,cluster_cols = F)
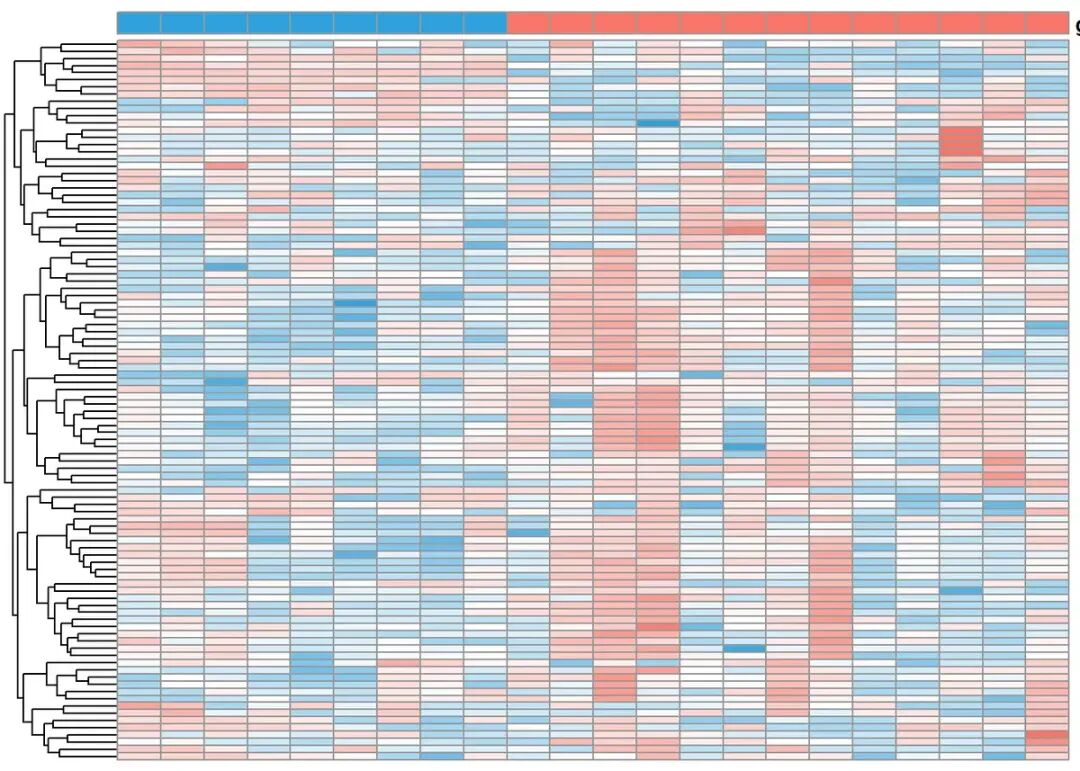
image.png
解决办法4
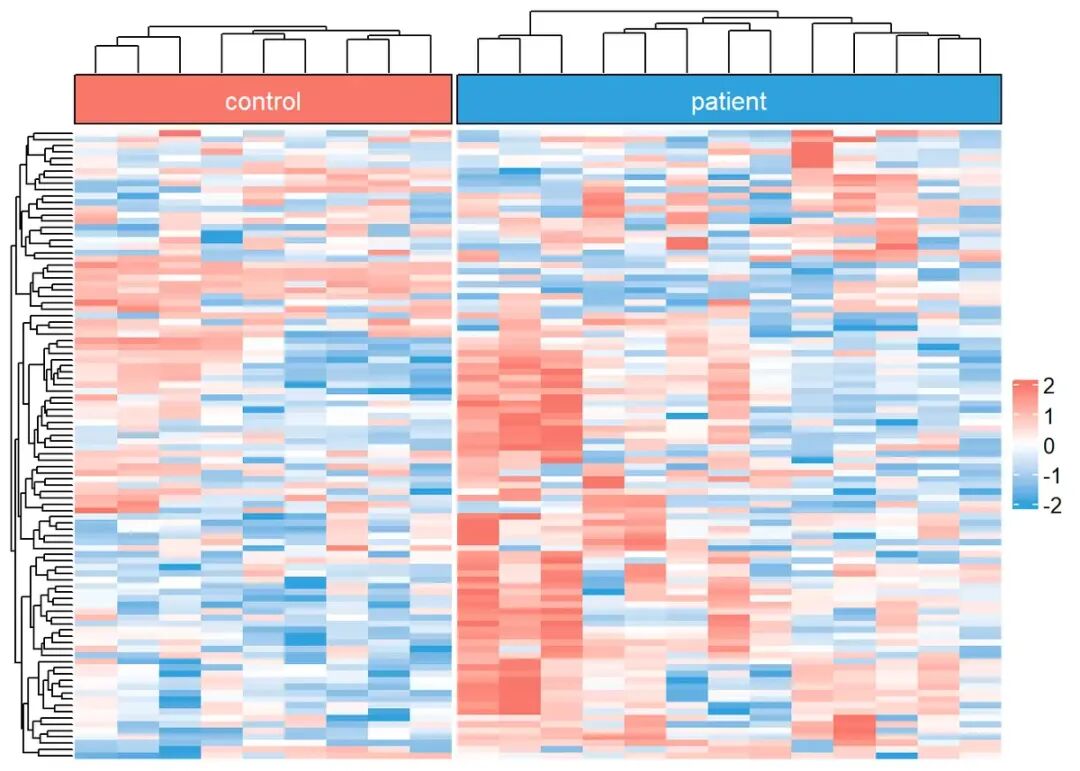
设置列不聚类是可以的,不过,当样本数量多起来的时候,组内的数据规律就无法清晰的展示。
还可以考虑组内聚类这样的操作咯。这个只能用complexheatmap来实现,我还顺便找到了注释条加分组标签的画法,变得比常规热图好看一丢。
library(ComplexHeatmap)
library(circlize)
col_fun = colorRamp2(c(-2, 0, 2), c("#2fa1dd", "white", "#f87669"))
top_annotation = HeatmapAnnotation(
cluster = anno_block(gp = gpar(fill = c("#f87669","#2fa1dd")),
labels = levels(Group),
labels_gp = gpar(col = "white", fontsize = 12)))
m = Heatmap(t(scale(t(exp[g,]))),name = " ",
col = col_fun,
top_annotation = top_annotation,
column_split = Group,
show_heatmap_legend = T,
border = F,
show_column_names = F,
show_row_names = F,
use_raster = F,
cluster_column_slices = F,
column_title = NULL)
m
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2026-02-02,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录


