这半吊子的公共数据把我的学生给欺负了
0.问题
接到学生求助说画pca图出问题了,如下:
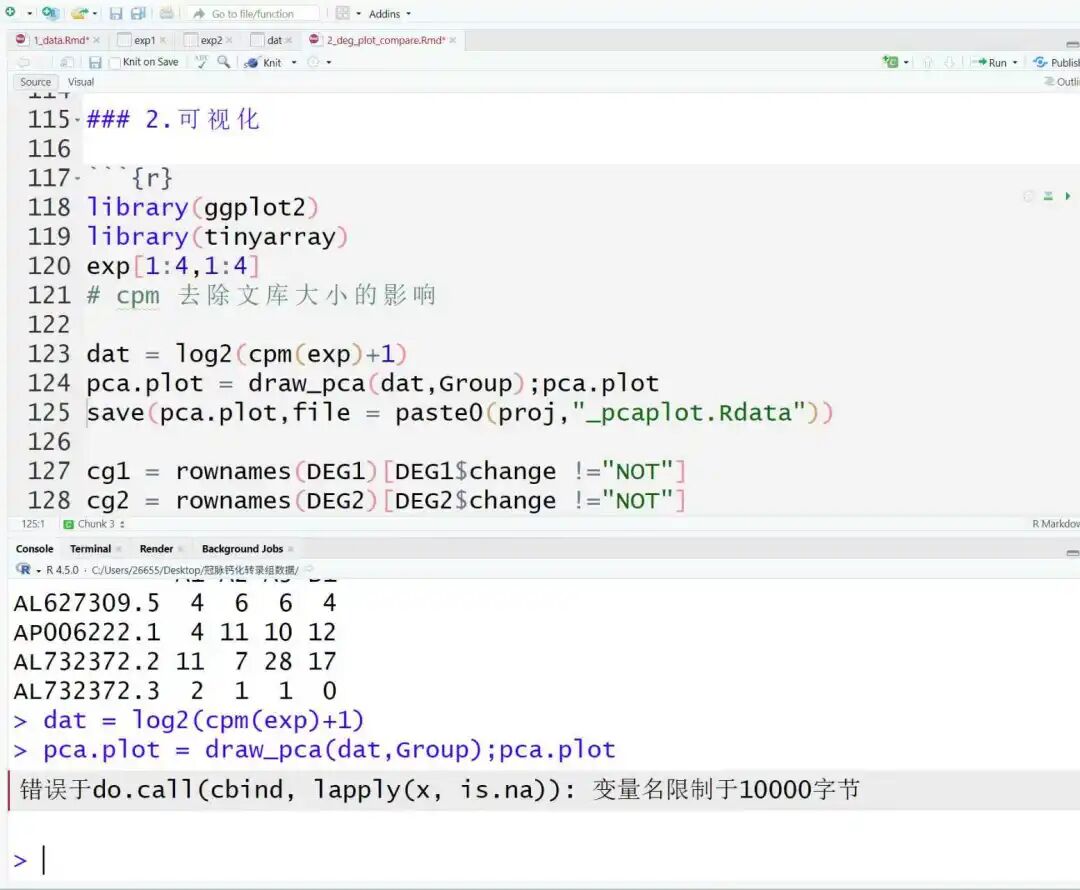
我要了他的dat和Group数据的截图,没有发现问题。于是要了他的压缩包

此时我的怀疑是我的函数有问题,心虚.jpg!
1.开始排查
1.1 排除函数问题
我把函数的源代码拆出来运行,发现这个报错出自PCA函数,就是计算PCA的时候报错。好了,分分钟排除掉了自己的嫌疑!哈哈哈哈。
dat = as.data.frame(t(exp))
dat.pca <- FactoMineR::PCA(dat, graph = FALSE)
1.2 锁定基因名的问题
报错信息指向变量名(机器学习里的变量,此处是基因),那就改名试试。我运行了:
colnames(dat) = paste0("a",1:ncol(dat))
再运行PCA就成功了。所以一定是基因名有问题。
但是基因名能有啥问题呢?看表达矩阵的样子是正常的啊。
1.3 检查基因名
特殊字符?空格?重复值?编码方式?都排查一遍了,没发现问题。
再看一眼报错信息,提到了大小,那就是基因名字的字数太多了?
好家伙,还真是!
> head(sort(str_length(colnames(dat)),decreasing = T))
[1] 12575 5199 5199 25 23 22
什么基因名能有一万多个字符啊!
2.问题溯源
往前追溯代码和数据,发现在数据读取一进来的时候,他的基因名称列天生就是有问题的。所以并不是学生在处理的时候搞错了,这作者坑人啊!洗清学生嫌疑。
dat = read.delim("GSE254862_featureCountsPhenotypic.txt.gz",
check.names = F)
k = !duplicated(dat$Geneid);table(k)
## k
## FALSE TRUE
## 2 61908
library(dplyr)
library(stringr)
dat = distinct(dat,Geneid,.keep_all = T)
#排查geneid的字符串长度,发现有3个异常
head(sort(str_length(dat$Geneid),decreasing = T))
## [1] 12575 5199 5199 25 23 22
dat$Geneid[which.max(str_length(dat$Geneid))]#刷屏警告
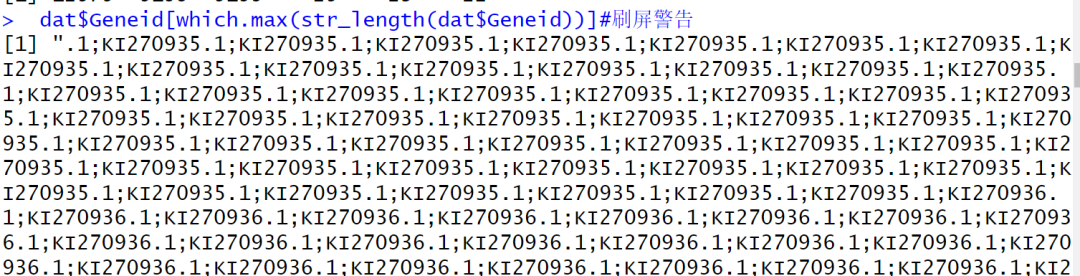
说实话,我确实不知道这函数对基因名称长度有限制,不遇到这个坑怎么能知道。
3.修正后的数据整理代码
他这个数据除了基因长度有问题外,还有一个毛病,就是基因名称是杂交的,一半是symbol,一半是ensemblid。
所以我一并写出整理的代码吧,真是个特殊的数据。
1.起个项目名字
TCGA的数据,统一叫TCGA-xxxx,非TCGA的数据随意起名,不要有特殊字符即可。 ⭐
rm(list=ls())
proj = "GSE254862"
2.读取和整理数据
2.1 表达矩阵
dat = read.delim("GSE254862_featureCountsPhenotypic.txt.gz",
check.names = F)
k = !duplicated(dat$Geneid);table(k)
#> k
#> FALSE TRUE
#> 2 61908
library(dplyr)
library(stringr)
dat = distinct(dat,Geneid,.keep_all = T)
#排查geneid的字符串长度,发现有3个异常
head(sort(str_length(dat$Geneid),decreasing = T))
#> [1] 12575 5199 5199 25 23 22
# dat$Geneid[which.max(str_length(dat$Geneid))]#刷屏警告
#观察发现它们以;为分隔符,包含了很多个重复的基因名称
#方法1:去掉这样的行
#k2 = str_detect(dat$Geneid,";");table(k2)
#dat = dat[!k2,]
#方法2:只留第一个id
dat$Geneid = str_split_i(dat$Geneid,";",1)
exp = as.matrix(tibble::column_to_rownames(dat,"Geneid"))
2.2 临床信息
clinical = 1 #⭐有临床信息就读取,没有临床信息就写1,占位置,不用改后面代码
3.表达矩阵行名ID转换
trans_exp_new 仅用于ensemble 转symbol
ke = str_starts(rownames(exp),"ENS");table(ke)
#> ke
#> FALSE TRUE
#> 39651 22257
library(tinyarray)
exp1 = trans_exp_new(exp[ke,])
#> Warning in AnnoProbe::annoGene(rownames(exp), ID_type = "ENSEMBL", species =
#> species): 11.56% of input IDs are fail to annotate...
exp2 = exp[!ke,]
exp = rbind(exp1,exp2)
exp = exp[!duplicated(rownames(exp)),]
exp=exp[,c(1:3,10:12)] #学生表示只想要这几个样本
colnames(exp)
#> [1] "A1" "A2" "A3" "B1" "B2" "B3"
4.基因过滤
需要过滤一下那些在很多样本里表达量都为0或者表达量很低的基因。过滤标准不唯一。
过滤之前基因数量:
nrow(exp)
#> [1] 59327
常用过滤标准1:
仅去除在所有样本里表达量都为零的基因
exp[is.na(exp)] <- 0
exp1 = exp[rowSums(exp)>0,]
nrow(exp1)
#> [1] 26862
常用过滤标准2(推荐):
仅保留在一半以上样本里表达的基因
exp = exp[apply(exp, 1, function(x) sum(x > 0) > 0.5*ncol(exp)), ]
nrow(exp)
#> [1] 18857
5.分组信息获取
TCGA的数据,直接用make_tcga_group给样本分组(tumor和normal),其他地方的数据分组方式参考芯片数据pipeline/02_group_ids.R
⭐ 改分组信息
colnames(exp)
#> [1] "A1" "A2" "A3" "B1" "B2" "B3"
library(stringr)
Group = ifelse(str_detect(colnames(exp),"A"),"A","B")
Group = factor(Group,levels = c("A","B"))
table(Group)
#> Group
#> A B
#> 3 3save(exp,Group,proj,clinical,file = paste0(proj,".Rdata"))
这次画图就不会报错啦。
exp = log2(edgeR::cpm(exp)+1)
draw_pca(exp,Group)

本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-12-01,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录


