CUT&Tag 数据处理和分析教程(7)
CUT&Tag 数据处理和分析教程(7)

数据科学工厂
发布于 2025-04-23 11:20:56
发布于 2025-04-23 11:20:56
过滤
某些项目可能需要对比对质量分数进行更严格的过滤。本文细讨论了bowtie如何分配质量分数,并举例说明。
MAPQ(x) = -10 * log10log10(P(x is mapped wrongly)) = -10 * log10(p)
其范围从0到37、40或42。
使用samtools view -q minQualityScore命令将剔除所有低于定义的minQualityScore的对齐结果。
##== linux command ==##
minQualityScore=2
samtools view -q $minQualityScore ${projPath}/alignment/sam/${histName}_bowtie2.sam >${projPath}/alignment/sam/${histName}_bowtie2.qualityScore$minQualityScore.sam
文件格式转换
本节是为峰值调用和可视化做准备所需的内容,其中需要进行一些过滤和文件格式转换。
##== linux command ==##
## Filter and keep the mapped read pairs
samtools view -bS -F 0x04 $projPath/alignment/sam/${histName}_bowtie2.sam >$projPath/alignment/bam/${histName}_bowtie2.mapped.bam
## Convert into bed file format
bedtools bamtobed -i $projPath/alignment/bam/${histName}_bowtie2.mapped.bam -bedpe >$projPath/alignment/bed/${histName}_bowtie2.bed
## Keep the read pairs that are on the same chromosome and fragment length less than 1000bp.
awk '$1==$4 && $6-$2 < 1000 {print $0}' $projPath/alignment/bed/${histName}_bowtie2.bed >$projPath/alignment/bed/${histName}_bowtie2.clean.bed
## Only extract the fragment related columns
cut -f 1,2,6 $projPath/alignment/bed/${histName}_bowtie2.clean.bed | sort -k1,1 -k2,2n -k3,3n >$projPath/alignment/bed/${histName}_bowtie2.fragments.bed
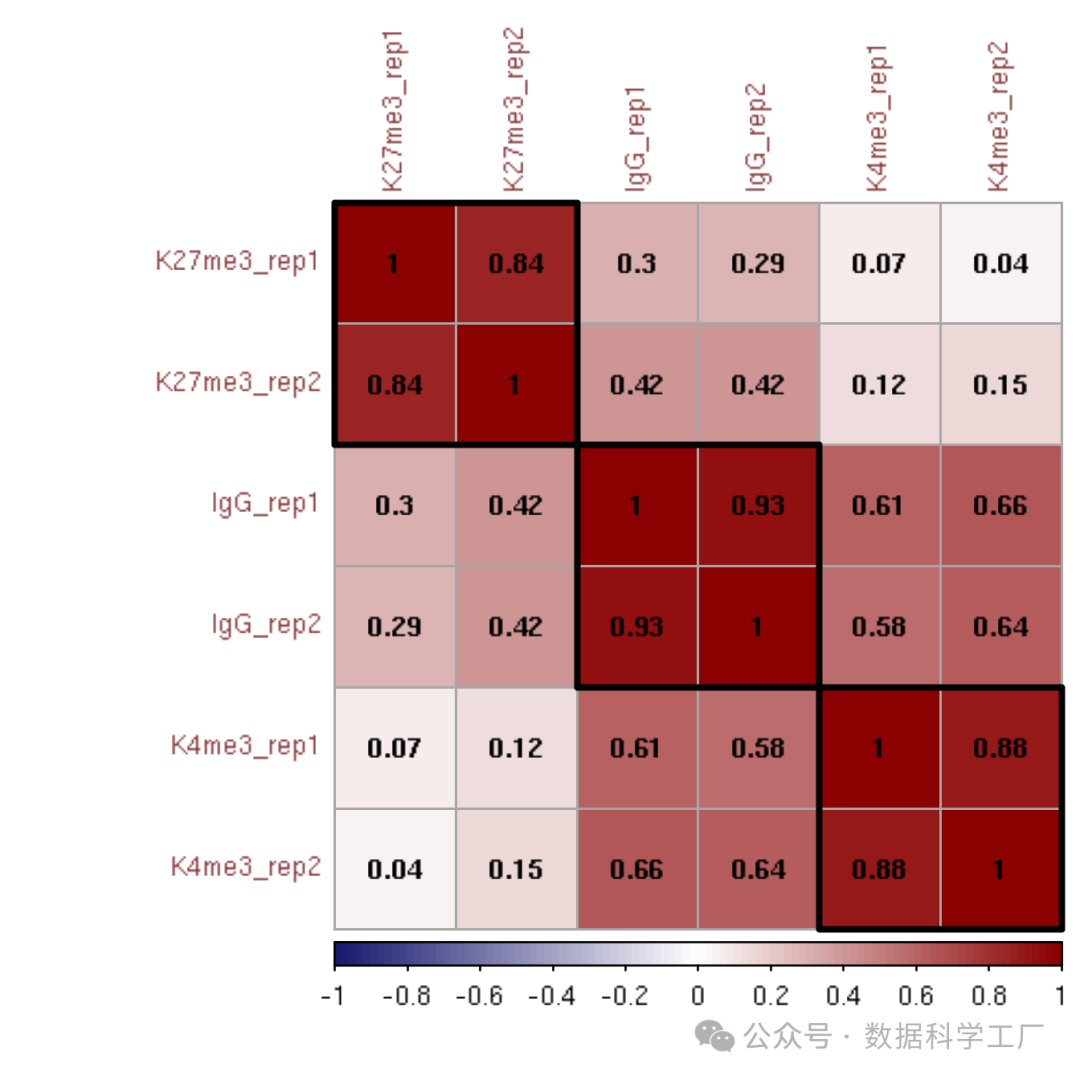
评估可重复性
为了研究重复样本之间以及不同条件下的可重复性,将基因组分成500 bp的片段,并计算每个片段中读取计数的log2转换值在重复数据集之间的皮尔逊相关性。多个重复样本和IgG对照数据集以层次聚类相关性矩阵的形式展示。
##== linux command ==##
## We use the mid point of each fragment to infer which 500bp bins does this fragment belong to.
binLen=500
awk -v w=$binLen '{print $1, int(($2 + $3)/(2*w))*w + w/2}' $projPath/alignment/bed/${histName}_bowtie2.fragments.bed | sort -k1,1V -k2,2n | uniq -c | awk -v OFS="\t" '{print $2, $3, $1}' | sort -k1,1V -k2,2n >$projPath/alignment/bed/${histName}_bowtie2.fragmentsCount.bin$binLen.bed
##== R command ==##
reprod = c()
fragCount = NULL
for(hist in sampleList){
if(is.null(fragCount)){
fragCount = read.table(paste0(projPath, "/alignment/bed/", hist, "_bowtie2.fragmentsCount.bin500.bed"), header = FALSE)
colnames(fragCount) = c("chrom", "bin", hist)
}else{
fragCountTmp = read.table(paste0(projPath, "/alignment/bed/", hist, "_bowtie2.fragmentsCount.bin500.bed"), header = FALSE)
colnames(fragCountTmp) = c("chrom", "bin", hist)
fragCount = full_join(fragCount, fragCountTmp, by = c("chrom", "bin"))
}
}
M = cor(fragCount %>% select(-c("chrom", "bin")) %>% log2(), use = "complete.obs")
corrplot(M, method = "color", outline = T, addgrid.col = "darkgray", order="hclust", addrect = 3, rect.col = "black", rect.lwd = 3,cl.pos = "b", tl.col = "indianred4", tl.cex = 1, cl.cex = 1, addCoef.col = "black", number.digits = 2, number.cex = 1, col = colorRampPalette(c("midnightblue","white","darkred"))(100))
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-04-22,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

