使用Microeco绘制微生物共现网络并指定节点颜色
网络图在科研论文中非常常见。
基于微生物组数据绘制Co-occurence network的方法网上已有非常多的教程,但在试过多种方法以后,我发现还是R包 microeco最简单,再加上Gephi进行美化一般能做出可用于发表的图。
Microeco的中文介绍可以参照这篇文章。
本期我们主要解决基于Gephi生成的多个网络图颜色不统一的问题。
示例数据获取
本期的示例数据来自microeco
library(microeco)
library(magrittr)
library(ggplot2)
data("sample_info_16S") # 分类表
data("otu_table_16S") # OTU丰度表
data("taxonomy_table_16S") # 物种注释表
dataset <- microtable$new(sample_table = sample_info_16S,
otu_table = otu_table_16S,
tax_table = taxonomy_table_16S)
dataset
使用microeco制作igraph对象
dataset$tax_table %<>% subset(Kingdom == "k__Bacteria")
dataset$filter_pollution(taxa = c("mitochondria", "chloroplast"))
dataset$tidy_dataset()
## 当OTU数目比较多时候,使用R包WGCNA提代R中基础函数计算相关性
# 加载WGCNA包
t1 <- trans_network$new(dataset = dataset,
cal_cor = "WGCNA", # 使用WGCNA包计算相关性
taxa_level = "OTU", # 选择分类水平,可选OTU, Genus, Family, Order, Class, Phylum, Kingdom
filter_thres = 0.001, # 相对丰度过滤阈值
cor_method = "spearman") # 相关性计算方法
# 构建网络,需要igraph包
t1$cal_network(p_thres = 0.05,
COR_cut = 0.6,
add_taxa_name = "Kingdom",
delete_unlinked_nodes = TRUE,
COR_optimization = FALSE) # use random matrix theory (RMT) based method to determine the correlation coefficient
# 返回t1$res_network
t1$cal_module(
method = "cluster_fast_greedy",
module_name_prefix = "M")
如果你不需要对不同分类地位如Phylum或不同模块指定颜色,那么在这一步你就可以保存.gexf文件并打开Gephi进行美化了。
定义模块颜色
接下来有一个很重要的需求:将不同的模块/门分配给特定的颜色,以便在多个网络图中保持一致。
为什么有这样的需求?
- Gephi自带的色板颜色不算特别好看;
- Gephi无法将同一色板应用于多个网络图。
这对绘制组图来说非常重要。
(想必你也不想每张图都去AI绘制一个单独的图例吧?)
Gephi其实有两个插件可以用于分配颜色——"Give Colors To Edges" 和 "Give Colors To Nodes"。
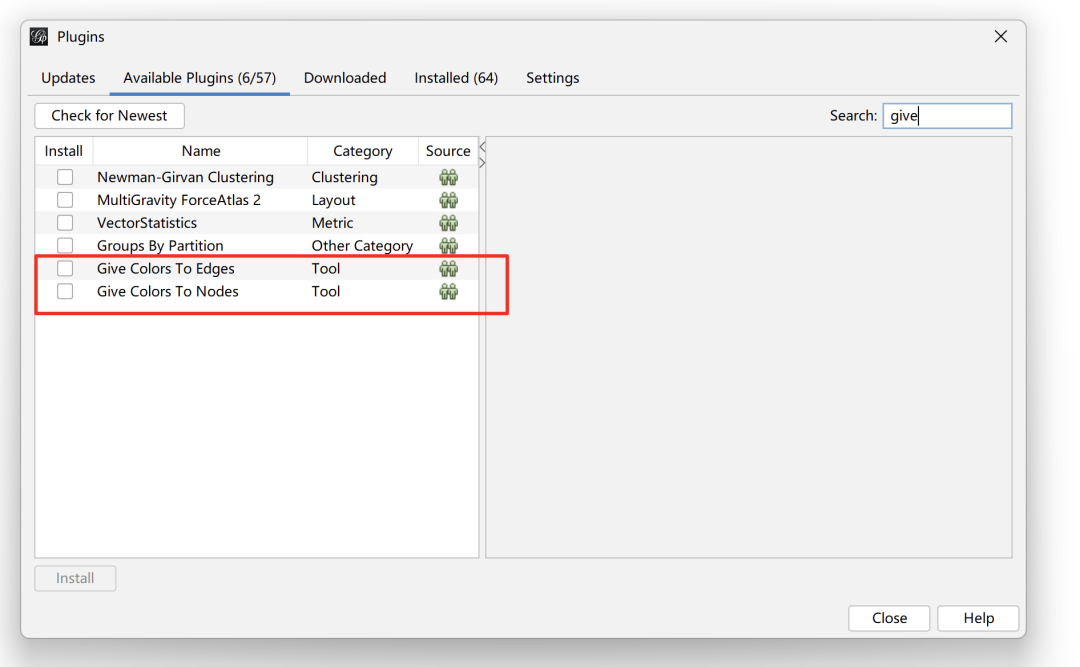
因此,在生成igraph对象以后,将颜色信息添加到igraph对象中,再使用插件即可指定颜色。
具体操作如下:
1 定义模块颜色映射
下列操作仅为示例
# 定义模块颜色映射
module_colors <- c("M1" = "#38678E",
"M2" = "#7935F6"
# 继续添加其他模块及其颜色映射
)
# 定义其他模块的颜色
other_color <- "#ff6347"
# 将颜色信息添加到igraph对象中
V(t1$res_network)$color <- ifelse(V(t1$res_network)$module %in% names(module_colors),
module_colors[V(t1$res_network)$module],
other_color)
t1$save_network("test.gexf")
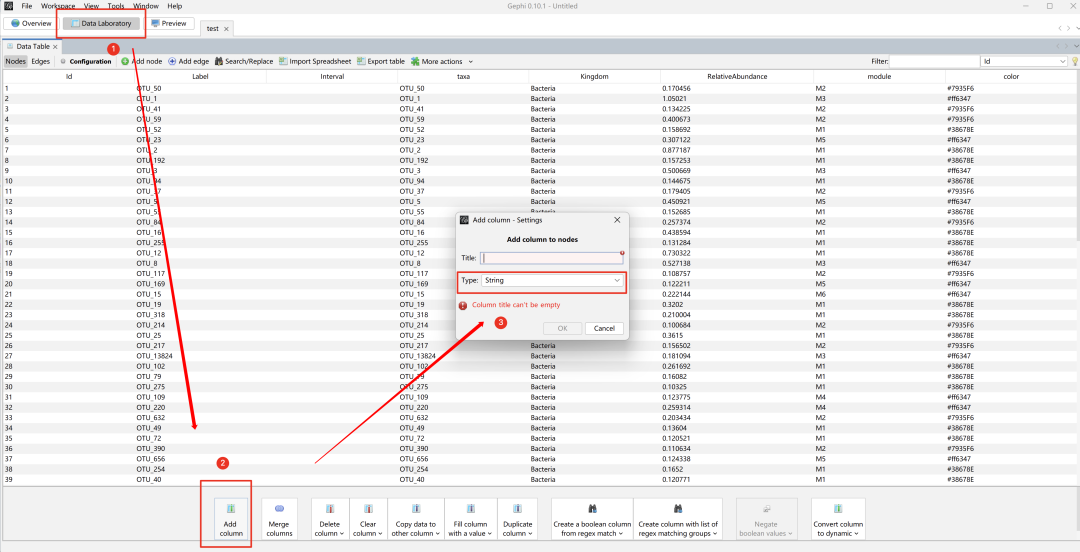
2 新增String列并复制颜色列
在Gephi中打开"test.gexf",在Data Laviratory中,新增一列名为"colour"以区分在R中定义的color。
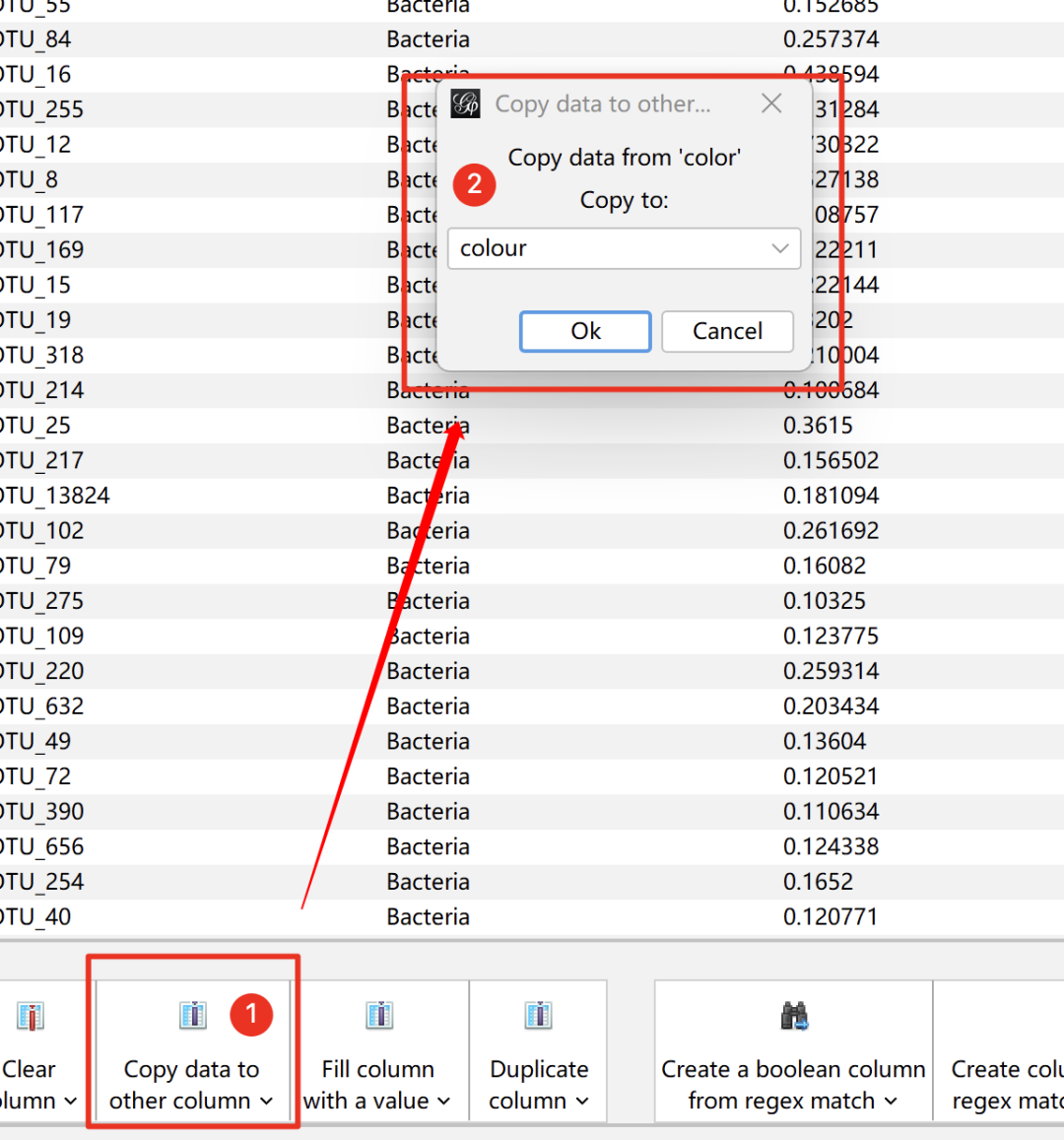
接下来,点击下方的"Copy data to other column"。
3 点击"Give Colors To Nodes"
点击"Give Colors To Nodes"后插件自动识别对应颜色,最终的网络图如图所示。
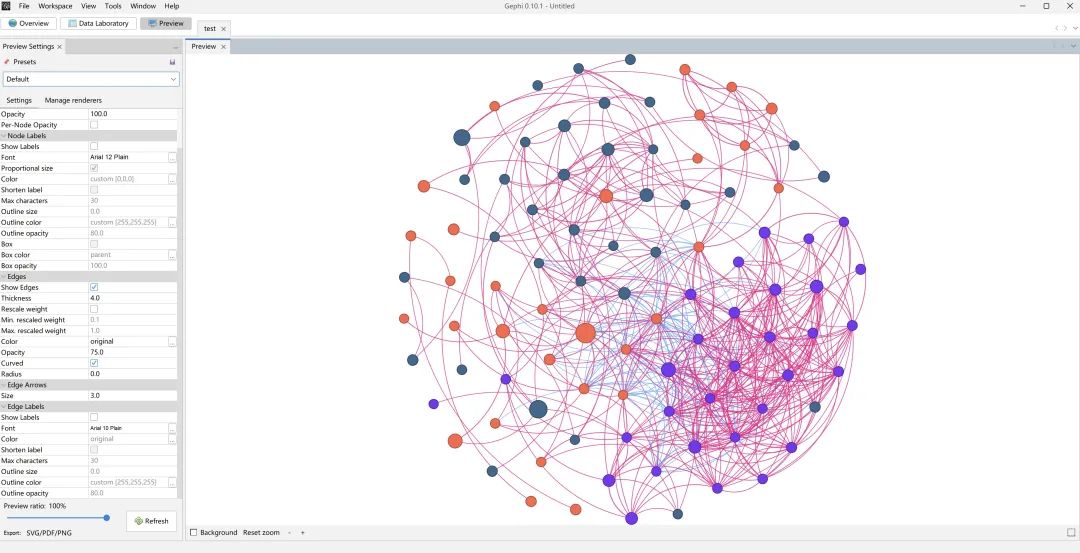
给边指定颜色也是同样的思路。
汇总代码
各位请根据需求修改
library(microeco)
library(magrittr)
library(ggplot2)
theme_set(theme_bw())
data("sample_info_16S") # 分类表
data("otu_table_16S") # OTU丰度表
data("taxonomy_table_16S") # 物种注释表
dataset <- microtable$new(sample_table = sample_info_16S,
otu_table = otu_table_16S,
tax_table = taxonomy_table_16S)
dataset
dataset$tax_table %<>% subset(Kingdom == "k__Bacteria")
dataset$filter_pollution(taxa = c("mitochondria", "chloroplast"))
dataset$tidy_dataset()
## 当OTU数目比较多时候,使用R包WGCNA提代R中基础函数计算相关性
# 加载WGCNA包
t1 <- trans_network$new(dataset = dataset,
cal_cor = "WGCNA", # 使用WGCNA包计算相关性
taxa_level = "OTU", # 选择分类水平,可选OTU, Genus, Family, Order, Class, Phylum, Kingdom
filter_thres = 0.001, # 相对丰度过滤阈值
cor_method = "spearman") # 相关性计算方法
# 构建网络,需要igraph包
t1$cal_network(p_thres = 0.05,
COR_cut = 0.6,
add_taxa_name = "Kingdom",
delete_unlinked_nodes = TRUE,
COR_optimization = FALSE) # use random matrix theory (RMT) based method to determine the correlation coefficient
# 返回t1$res_network
t1$cal_module(
method = "cluster_fast_greedy",
module_name_prefix = "M")
# 定义模块颜色映射
module_colors <- c("M1" = "#38678E",
"M2" = "#7935F6"
# 继续添加其他模块及其颜色映射
)
# 定义其他模块的颜色
other_color <- "#ff6347"
# 将颜色信息添加到igraph对象中
V(t1$res_network)$color <- ifelse(V(t1$res_network)$module %in% names(module_colors),
module_colors[V(t1$res_network)$module],
other_color)
t1$save_network("test.gexf")
背后故事
我不理解为什么Gephi这么多年了还不出一个legend的选项,后期从AI打开,再手动绘制legend的过程实在是太太太太麻烦了!
好在还有插件勉强能用。
在探索插件的过程中也有一个小插曲——为什么要在Data Laboratory中新建一列并复制color列呢?
通过R直接将color列写入igraph对象后,我们可以看到尽管颜色已经对应上了不同模块

但插件无法正确识别——"No String node attribute"。
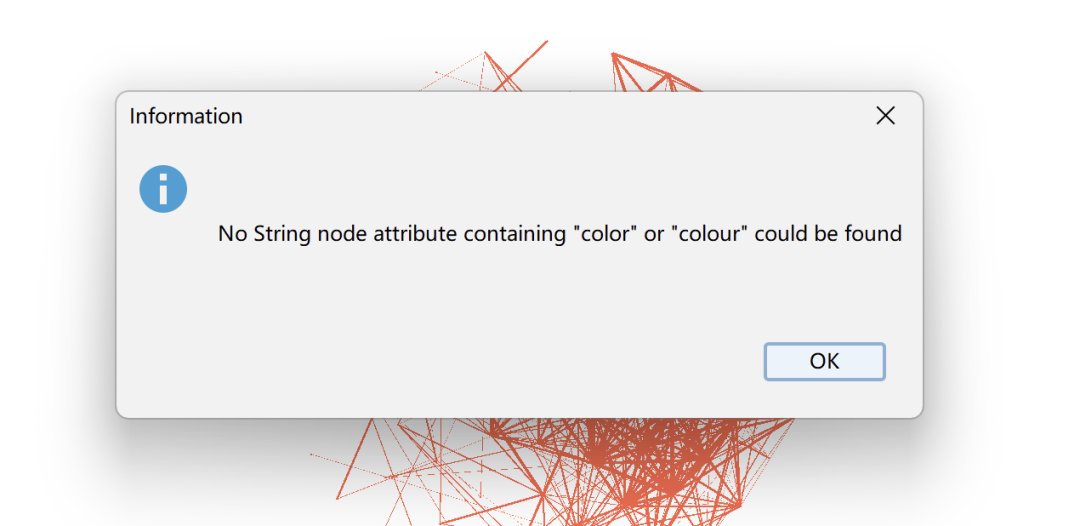
在R中查看生成的igraph对象的颜色信息,目前为character,可就算再使用toString()转换整列内容,也还是无法被"Give colors to nodes"识别,似乎R的string和Gephi的String就是无法兼容。

于是我在Gephi的github上写了一个issue[1]。

写完发现,Gephi的issue已经有很久没有人回应了...
于是转战寻找Give Colors To Nodes的作者,好在找到了作者Clement Levallois的Twitter,私信以后,他迅速给我解决了问题,点赞!!
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2024-04-05,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

